1 引言
新冠肺炎是一种全球性的流行病,严重威胁人类健康。截至2021年2月23日,全球已有192个国家和地区累计报告逾1.11亿例确诊病例,其中逾247.3万人死亡[1],是人类历史上致死人数最多的流行病之一。由于可能叠加其他肺部疾病,CT图像上的毛玻璃病变和结节不能作为确诊新冠肺炎的金标准[2],但其在病人的早期筛查以及愈后恢复等阶段都具有重要作用。在早期筛查阶段,CT比PCR(Polymerase chain reaction, PCR)更为敏感,可在PCR阳性前出现感染峰值,可更快速地识别出疑似病例并进行隔离,适合于患者较多的情形[3];在确认患者已确诊的情况下,我们可以通过不同时期CT图像中病灶的变化来定量地诊断患病程度和治疗效果;在用药痊愈的情况下,也可通过CT图像来观察病人的恢复程度[4]。
CT(Computed Tomography)肺部图像的毛玻璃病变是检测新冠肺炎的重要指标之一,准确分割病变区域对诊断、治疗和预后有重要意义。近年来,以U-net[5]为基本框架的CNN(Convolutional Neural Network)模型已成CT图像病灶分割的主流算法[6]。然而,作为世界性的流行性疾病,新冠肺炎患者的病灶分割仍然存在较多困难。一方面,由于CT图像中新冠肺炎病灶大小和位置变化较大,比如肺实变(Consolidation)很小且与周围体素相差较小,容易导致模型的假阴[7-8];另一方面,不同设备的CT扫描参数存在差异,导致数据存在明显异质性,为病灶分割带来困难。目前,已有学者针对这些问题开展了相关研究。Qi等人对中国境内多所医院的小规模病例使用机器学习模型进行特征回归[9],获得了较好的效果;Gao等人使用分类和分割的多任务注意力模块来减少不同成像参数的影响,在一定程度上改善了数据异质性导致的算法性能下降[10]。由于医学影像标注的缺乏,基于机器学习的方法只能在小数据集上进行[9],但使用数据驱动的卷积网络模型容易发生过拟合。为解决该问题,Oulefki等人提出采用Kapur熵阈值对病灶进行分割的机器学习模型[11];同时,针对缺乏数据标注的应用场景,Fan等人提出采用基于注意力模块的卷积神经网络对病灶进行分割[7],也有研究使用预训练模型对有限标注的数据集进行小样本学习以提升分割效果[12]。
总之,从已公开发表的文献看,现有研究大多针对多站点背景下的小规模数据集开展,对大规模跨国多站点数据集进行新冠肺炎病灶分割的相关研究还较为少见。然而,基于新冠肺炎的全球流行性,对大规模跨国多站点的数据集进行病灶分割等研究极为重要和紧迫,具有现实的临床意义。
2 网络框架
2.1 总体框架
针对新冠肺炎的病灶分割问题,本文提出基于深度卷积神经网络的分割方法,其总体框架如图1所示。分割网络分为预处理模块、骨架网络以及集成学习三部分。预处理部分首先对原始的CT图像在Z轴进行重采样,再进行数据扩增;由于数据层厚并不稀疏,且特征差异明显,因此本文对扩增后的数据集取三维体素补丁,使得网络在三个维度进行参数共享,从而更准确地学习到各维度的差异;同时,由于输入图像在三个维度上的尺寸可能会有差异,需使用固定大小的体素补丁让模型能兼容XY平面上不同尺寸的输入图像。由于前景背景的比例不均衡,还可在截取补丁时使用ROI(Region of Interesting)提取来保持正负样本的比例,使模型在类别不均衡的CT图像中更好地学习图像特征。
在手工标注情形下,不同医生对CT图像的阅片侧重点不同,如有些医生侧重于整体,而有些则着重细节。为了适应于不同的阅片需求,本文使用基于3D U-net的改进网络[13]作为骨架网络,将截取出来的体素补丁分别输入高分辨率和低分辨率(两个感受野不同)骨架网络进行训练,同时学习肺部CT图像的整体和细节特征。下面对分割网络进行详细阐述。
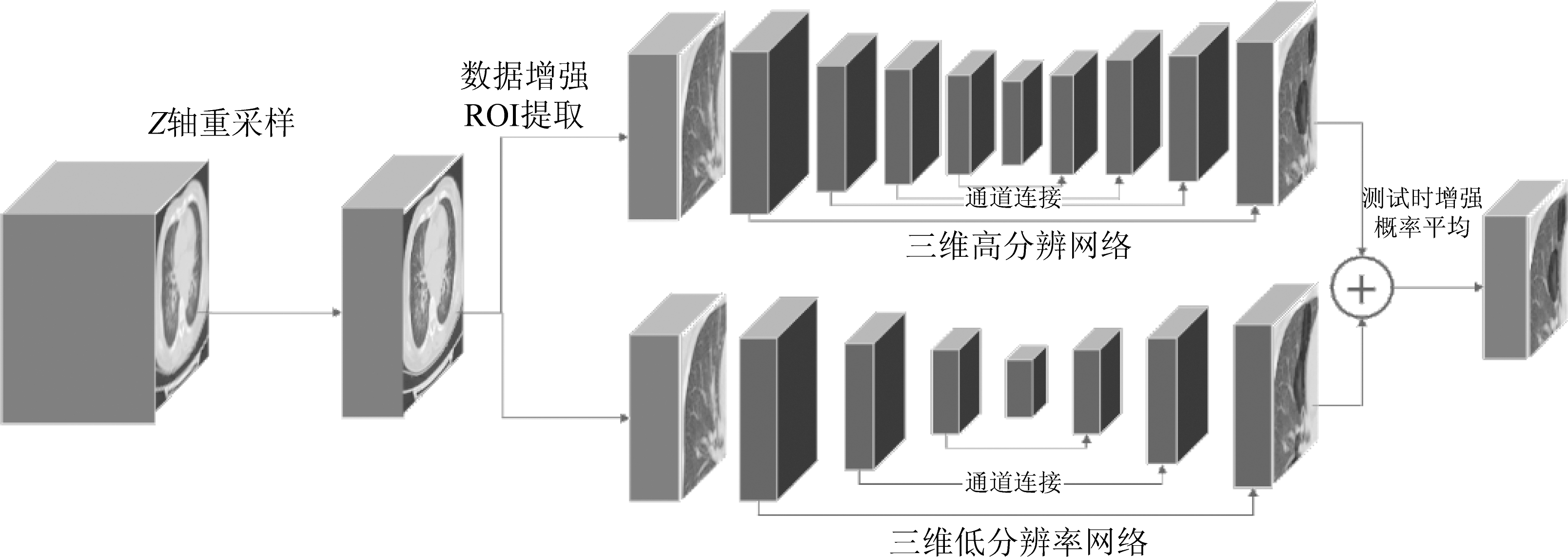
图1 网络总体框架
Fig.1 Overall framework of the network
2.2 预处理
预处理的重点在于如何解决输入图像中类别不均衡以及输入图像异质性的问题,包括Z轴重采样、数据扩增以及三维体素补丁ROI提取三部分。Z轴是CT图像的层厚维度,也是数据集中像素点间距变化最大的维度。尽管三维深度卷积网络在层厚维度上参数共享,但由于医学图像的数据量极小,某维度间距差异较大时使用三维体素补丁较难学习到该维度中隐含的有用模式,从而导致分割性能的下降。针对此问题,本文对其进行标准化重采样以提升维度学习的有效性。重采样的目标间距为训练集所有样本间距的百分之十,体素尺寸可重新计算如下:
Vtarget=(SsourceStarget)*Vsource
(1)
其中,Vsource和Ssource表示原来的体素尺寸和体素间距,Vtarget和Starget表示重采样后的体素尺寸和体素间距。Z轴的体素尺寸与体素间距的乘积为对应物理空间中的真实尺寸,从原间距变为目标间距后,需同时修改体素尺寸使其对应物理空间中的真实大小保持不变。
数据扩增方法用来缓解医学图像数据量偏少的问题。本文采用的数据扩增方法包括自然图像处理中常用的缩放旋转、亮度增强、高斯噪声、伽马校正等,也包括医学图像中常用的镜像翻转。考虑到多站点的CT图像中病灶大小多变且不均匀,给网络学习带来困难,因此本文的数据扩增方法中不采用弹性变换等数据扩增方法,以避免数据泄漏。最后,在预处理阶段,我们通过对扩增后的数据提取ROI来缓解类别不平衡的问题,即在提取三维体素补丁时以一定比例截取前景病灶区,使得随机裁剪的前景背景体素数量保持平衡。
2.3 骨架网络
本文方法基于3D U-net的改进模型[13],分别采用了低分辨率网络和高分辨率网络两个同体系的骨架网络。低分辨率网络比高分辨率网络的编解码层数少,分割的语义层级较低。批归一化将每个批次的数据归一化为带有可学习参数的正态分布,在减少数据分布偏移的同时加快神经网络的收敛,是目前常用的标准化方法;而实例归一化相比批归一化方法去掉了批次和通道,对每个样本的每个通道都单独计算可学习的参数,虽然加大了计算的成本,但可有效提升分割效果,且可以保持同一批次中不同样本以及特征图中不同通道之间的独立性。基于此,本文在网络中使用实例归一化。另外, Leaky ReLu与Relu函数相比,在X坐标负轴定义了一个较小的非零斜率α(一般可设为0.01),防止神经元在负值的时候不被激活,导致参数停止更新。因此,我们采用Leaky ReLu作为网络中的激活函数。其定义如下:
(2)
此外,骨架网络使用了深监督技术,在解码器的每一层都用1×1×1的三维卷积输出一个语义层级的分割结果,最后按分割结果的分辨率给予相应权重,分辨率越高则权重越高。对每个层级的分割结果分别计算损失,再将损失按权重进行加权获得最终损失。在该过程中由于综合考虑了不同语义层级的分割结果,使得训练出来的模型在各个语义层级上损失都较小,分割结果更加鲁棒。由于交叉熵损失函数LCE并不能全面地关注到前景和背景像素不均衡的问题,我们在网络中加入Soft Dice损失函数:
L=LCE+LDICE
(3)
其中,L代表模型整体的损失,LCE代表分割结果的交叉熵损失,LDICE代表分割结果的Soft Dice损失。Soft Dice损失将模型预测的softmax概率与真值标签逐元素相乘,使得原本作为性能指标的Dice可以作为损失函数参与梯度反向传播,减少了阈值化带来的信息损失。
2.4 集成学习
低分辨率网络和高分辨率网络的分割语义层级不同,即编解码器层数不同,将这两个网络的分割结果进行集成相当于在不同的语义层级上对输入图像进行分割。这种处理方法主要是由于输入的多站点CT图像不仅在Z轴存在差异,在XY平面上也存在差异。卷积网络作为一个局部的特征提取器,在对不同设置的输入图像进行特征提取时,不同语义层级的特征起到的贡献并不一致。将高分辨率网络与低分辨率网络进行集成能够较好地解决输入图像中XY平面上的差异问题。
3 实验结果及分析
本文实验的数据集来自于COVID-19肺部CT病变分割挑战赛[14],训练集包括199个未进行数据增强的核酸检测(Reverse transcription polymerase chain reaction,RT-PCR)呈阳性的新冠肺炎患者,以新冠肺炎病变作为分割标签的胸部CT图像,而测试集为46例核酸检测(RT-PCR)阳性新冠肺炎患者。由于本数据集包含了多个站点的数据,站点间数据分布存在较大差异,导致预测结果存在较大误差。为了便于后续叙述,我们首先做了如下定义:定义训练集中出现过的样本所归属的数据分布为可见域,把训练集中未出现过的样本数据分布称为未见域。测试集中的例子有多个来源,包括23例与训练集部分数据位于同一数据分布的可见域图像和23例图像设置与训练集数据分布都不相同的COVID-19-AR[15](Chest Imaging with Clinical and Genomic Correlates Representing a Rural COVID-19 Positive Population)未可见域图像。
数据集由英伟达开发的新冠肺炎分割卷积神经网络模型进行自动分割作为标签的雏形,再由经美国放射学委员会认证的放射科医生在此基础上进行校正形成最终真值标签。上述两个过程会给数据集(包括训练集和测试集)引入相似的噪声。数据集对于毛玻璃样浑浊、小叶间隔增厚和肺实变等辨别新冠肺炎的病变特征都使用前景标签进行像素级标注,而对于正常的肺部器官等都使用背景标签进行标注。基于此定义,新冠肺炎的病灶分割是一个二分类问题。
3.1 数据集分布
训练集和测试集在XY平面上的尺寸均为512 mm,在Z轴尺寸上存在较大差异。训练集的样本在Z轴上集中分布在53~73 mm范围内,而测试集在Z轴上尺寸分布在53~127 mm范围内。训练集和测试集都有少量位于200~300 mm范围的离群样本;对于体素间距,训练集在XY平面上的间距大约为0.74 mm,在Z轴上的间距大约为5 mm;测试集在XY平面上的间距大约为0.97 mm,Z轴上的间距大约为3 mm和5 mm。由上述数据可看出,在多站点的背景下体素尺寸和间距的差异较大。
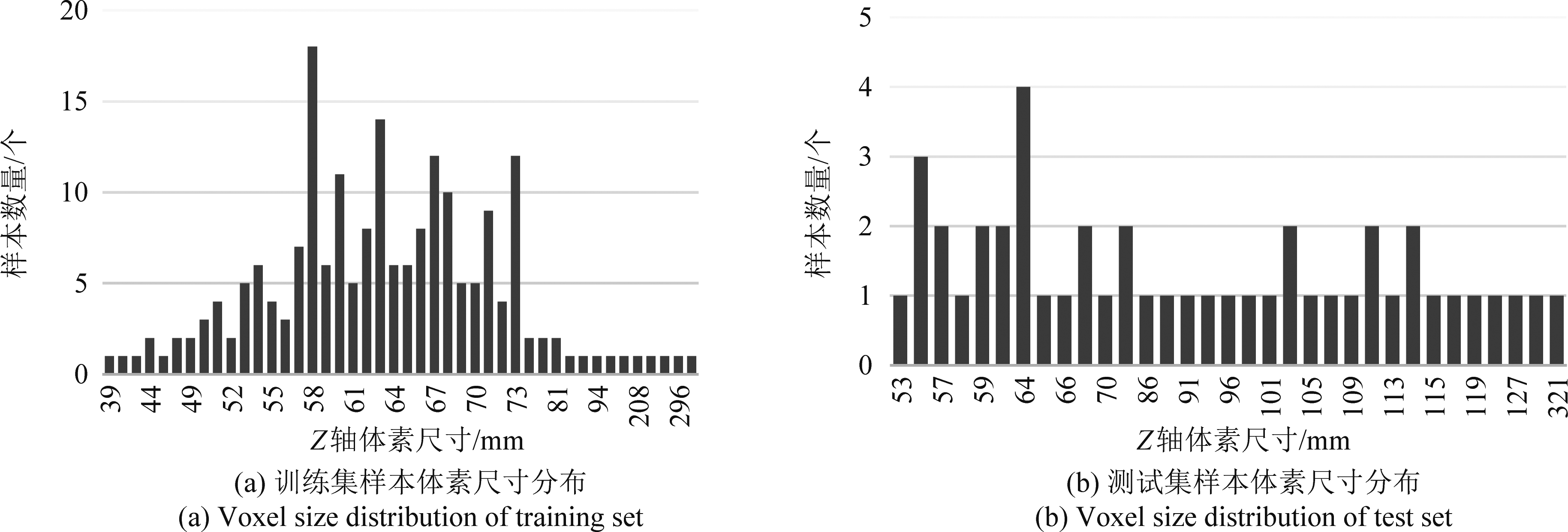
图2 数据集样本体素尺寸分布
Fig.2 Voxel size distribution of dataset
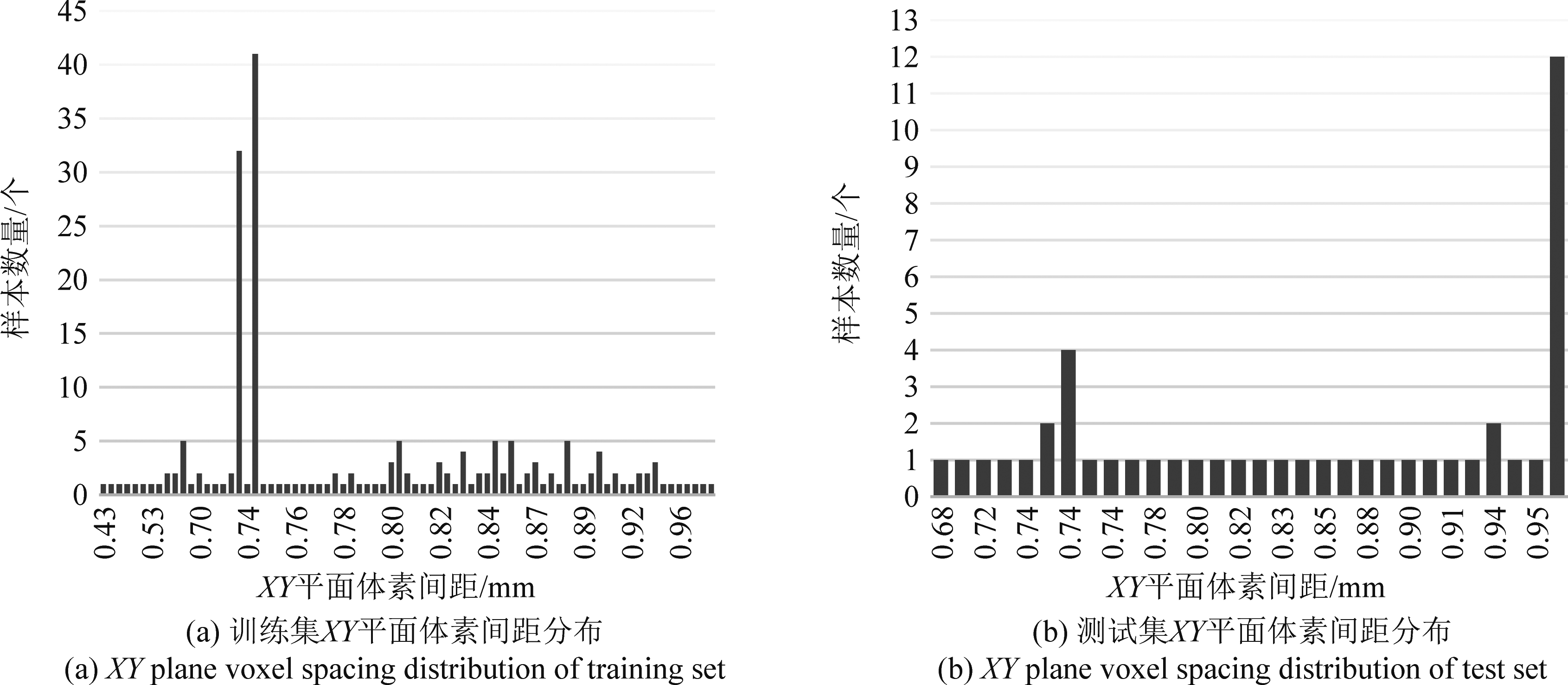
图3 数据集XY平面体素间距分布
Fig.3 XY plane voxel spacing distribution of dataset
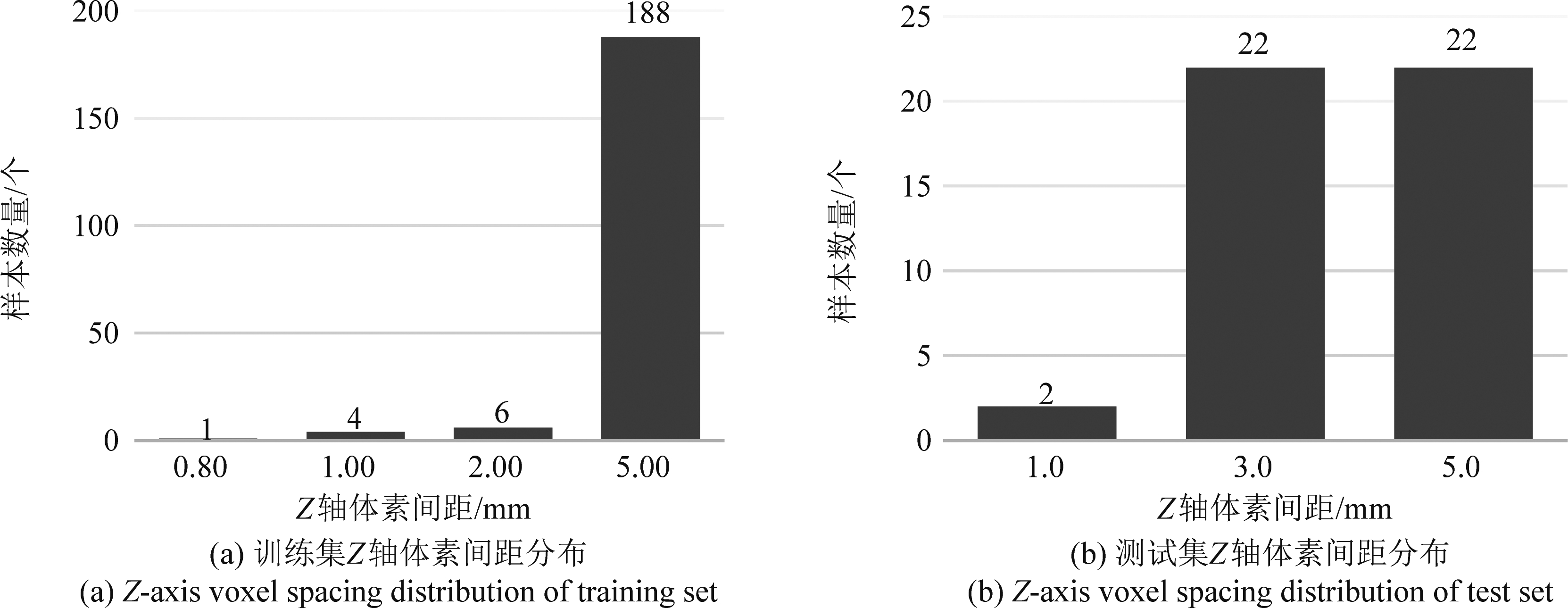
图4 数据集Z轴体素间距分布
Fig.4 Z-axis voxel spacing distribution of dataset
3.2 测试指标
本文采用三个指标评估算法分割性能:像素维度使用Dice,距离维度使用豪斯多夫距离,体积维度使用标准化的体积误差作为衡量模型性能的指标。Dice的定义为:
(4)
其中,A代表模型的预测集合,B代表真值标签,Dice为两个样本像素重叠比例的度量。
豪斯多夫距离的定义为:
dH(A,B)=max {dAB,dBA}= ![]()
(5)
对集合中的每个点,寻找与另外一个集合点的最短距离,然后对所有点的最短距离取最大值的95%,作为最终的豪斯多夫距离,目的是去除少量的离群值。标准化的体积误差定义为:
(6)
式中,将体积的误差除与真实标签的体积进行标准化。
3.3 测试集分割结果
实验中,我们用上述三个指标来评价比较本文提出模型和其他模型的性能。模型对测试集进行分割的结果如表1所示。从表中可以看出,使用2D切片作为输入的UNet由于并未学习到Z轴层厚的特征,在Dice指标上的表现最差,与其他以三维体素进行输入的模型相比存在较大差距;3D-UNet在豪斯多夫距离的指标上表现最优,但与3D-VNet以外的其他模型差异并不显著,其在Dice和标准化体积误差指标上表现一般;3D-VNet的结果与本文提出的模型较为相近,但由于使用了残差块结构,导致预测的结果豪斯多夫距离偏大。
表1 测试集分割指标
Tab.1 Indicators of test set
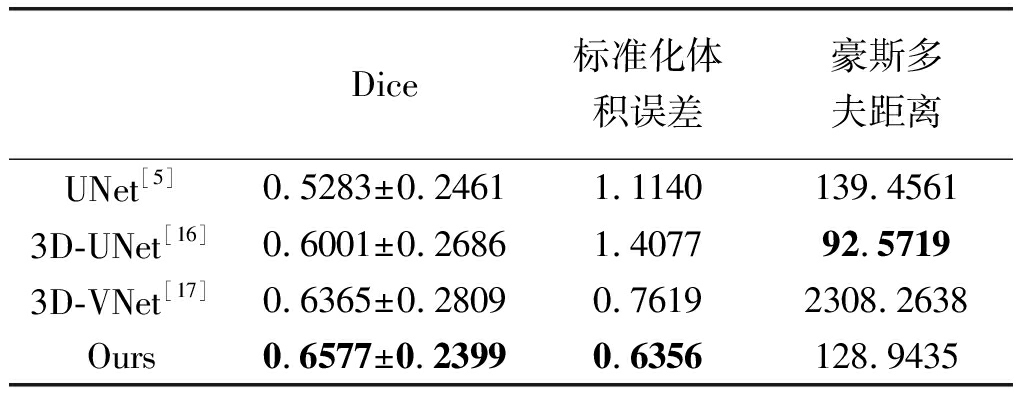
Dice标准化体积误差豪斯多夫距离UNet[5]0.5283±0.24611.1140139.45613D-UNet[16]0.6001±0.26861.407792.57193D-VNet[17]0.6365±0.28090.76192308.2638Ours0.6577±0.23990.6356128.9435
在基于上述三个指标的分析评价中,病灶分割出现遗漏或者过度分割均会导致病变体积的误差和Dice指标的变化。相对而言,分割的距离偏差不会造成重大影响。在综合平衡三个指标的情况下,分割的距离偏差可稍微放宽。从该角度分析,本文提出的模型在豪斯多夫距离中与其他模型的表现差异不大,但在较为重要的Dice指标以及标准化体积误差中表现优异。
我们将本文提出的模型与挑战赛指标前20名的其他模型进行了对比。这些模型可代表当前主流分割模型。各方法的分割结果如图5所示。对比其他算法,本文提出的算法对于测试集可见域样本的Dice与其他模型相差不大,但对于未见域样本进行分割时Dice更为优异,平均值位于全部20个模型的第2,箱线图的下端相比其他的算法也更好,说明本文提出的算法对于未见域样本的稳定性和泛化性更好,且模型对于测试集样本都有不错的分割效果。本文提出的模型最终获得了肺部CT病变分割挑战赛第8名的成绩。
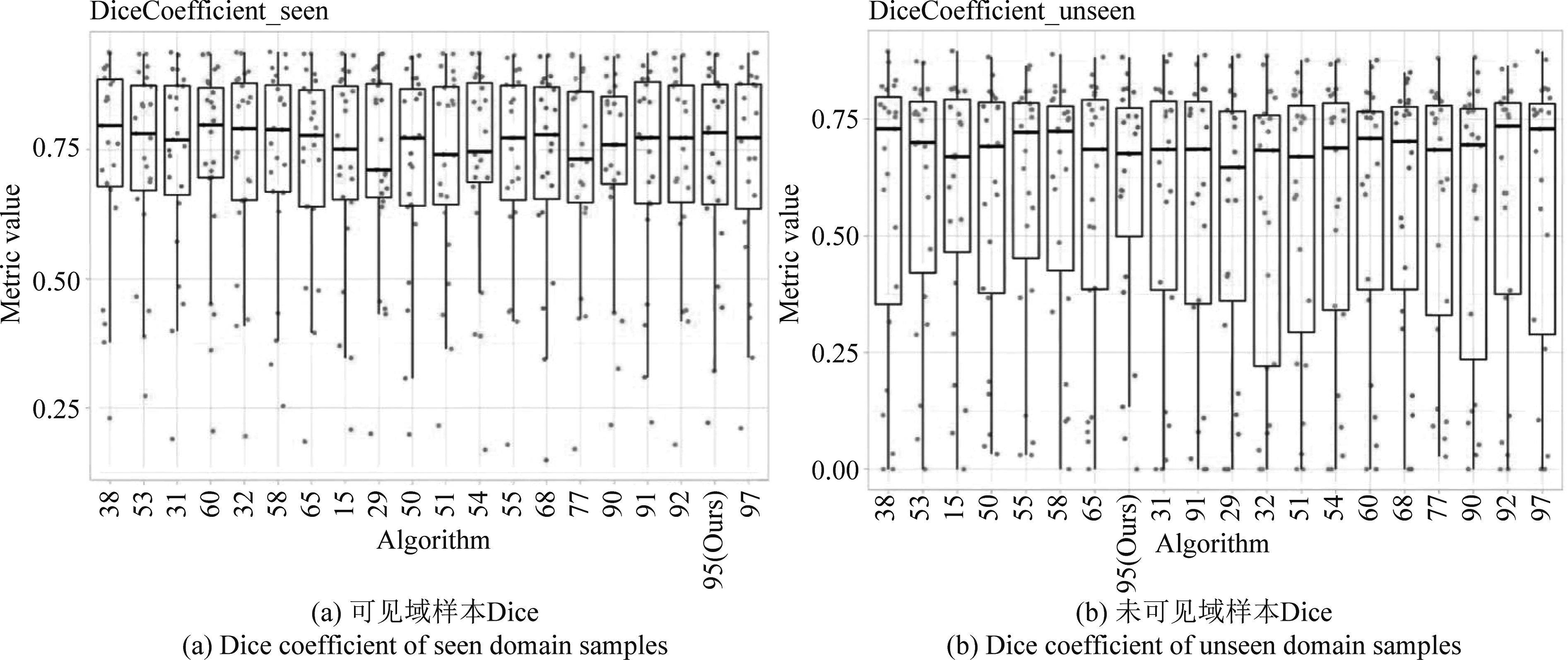
图5 与其他主流(State-of-the-art,SOTA)模型测试集Dice指标对比的箱线图(文中算法为95号算法)
Fig.5 Box plot of comparison with other SOTA model test sets in Dice metrics (ours algorithm is the No.95 algorithm)
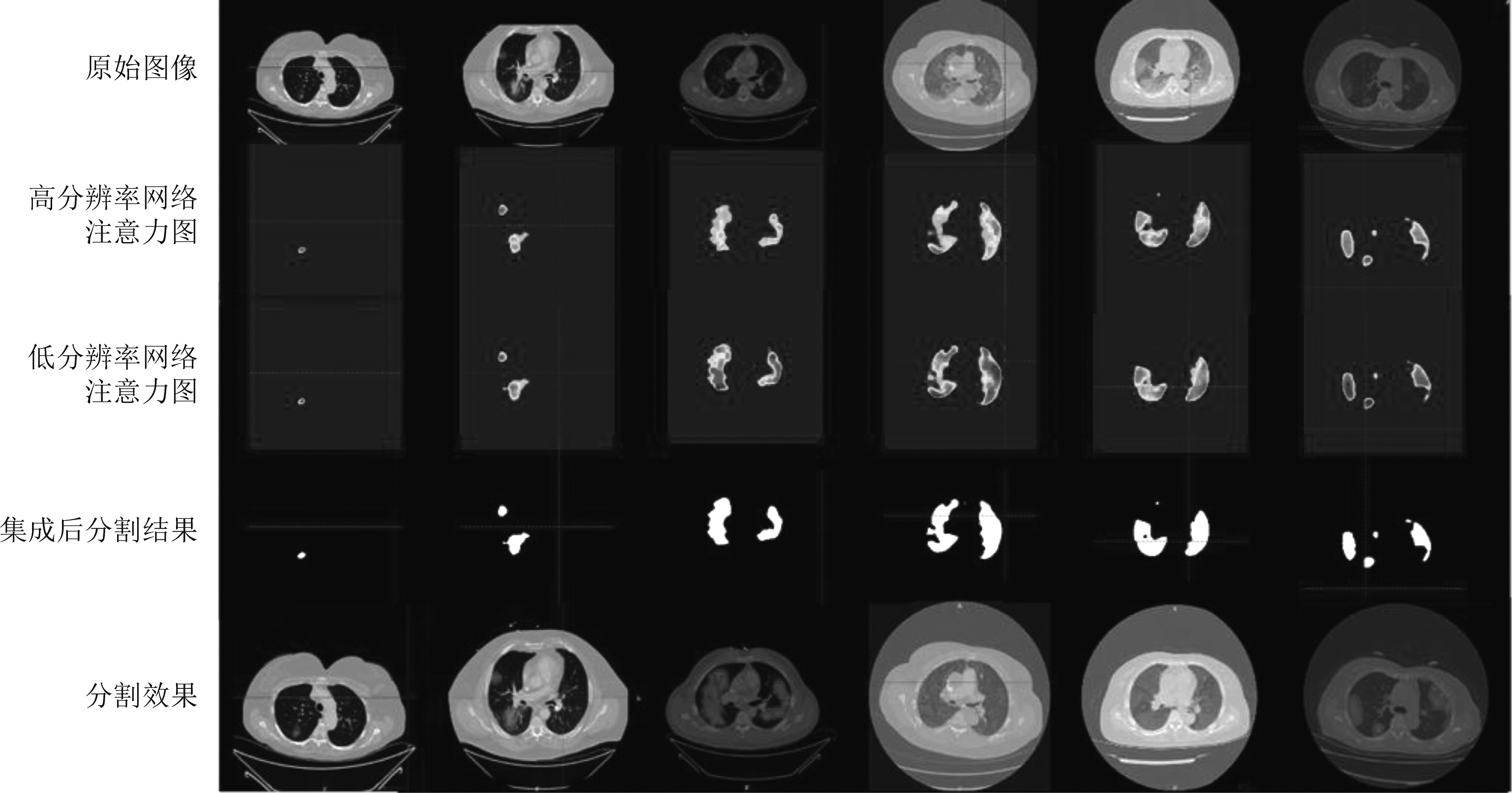
图6 测试集部分分割结果可视化
Fig.6 Visualization of test set segmentation results
图6为测试集中不同站点的部分样本可视化结果,我们采用Grad-CAM[18]类激活算法绘制了模型特征图的注意力图。可以看到三维高分辨率网络与低分辨率网络的注意力略有不同,将二者集成可有效实现不同分辨率情形下的优势互补。同时,从可视化结果中也可看出,我们的模型对于轻度或重度患者的新冠肺炎病灶进行有效的分割。
4 结论
本文对多站点新冠肺炎病灶分割问题进行了研究。针对多站点数据类别大小不均衡、图像尺寸和分辨率等设置差异较大的问题,使用重采样体素间距和ROI提取提供了较好的解决方案,并通过深监督和集成分割语义层级不同的网络来提升分割性能。同时在测试集上与挑战赛中前20名的网络模型进行了对比,从结果来看,我们提出的各个技术手段较为有效地提升了模型性能,在分割任务的各项指标上获得了不错的效果。
[1] ZHOU Longxi, LI Zhongxiao, ZHOU Juexiao, et al. A rapid, accurate and machine-agnostic segmentation and quantification method for CT-based COVID-19 diagnosis[J]. IEEE Transactions on Medical Imaging, 2020, 39(8): 2638-2652.
[2] WANG Guotai, LIU Xinglong, LI Chaoping, et al. A noise-robust framework for automatic segmentation of COVID-19 pneumonia lesions from CT images[J]. IEEE Transactions on Medical Imaging,2020,39(8):2653-2663.
[3] DONG E, DU H, GARDNER L. An interactive web-based dashboard to track COVID-19 in real time[J]. The Lancet Infectious Diseases, 2020, 20(5): 533-534.
[4] ROTZINGER D C, BEIGELMAN-AUBRY C, VON GARNIER C, et al. Pulmonary embolism in patients with COVID-19: Time to change the paradigm of computed tomography[J]. Thrombosis Research, 2020, 190: 58-59.
[5] RONNEBERGER O, FISCHER P, BROX T. U-net: Convolutional networks for biomedical image segmentation[C]∥Medical Image Computing and Computer-Assisted Intervention-MICCAI 2015, 2015: 234-241.
[6] 刘敏, 方志军, 高永彬. 多阶段冠状动脉CTA三维分割算法[J]. 信号处理, 2019, 35(11): 1911-1918.
LIU Min, FANG Zhijun, GAO Yongbin. Multi-stage three-dimensional segmentation algorithm for coronary CTA[J]. Journal of Signal Processing, 2019, 35(11): 1911-1918.(in Chinese)
[7] FAN Dengping, ZHOU Tao, JI Gepeng, et al. Inf-net: Automatic COVID-19 lung infection segmentation from CT images[J]. IEEE Transactions on Medical Imaging, 2020, 39(8): 2626-2637.
[8] SHI Feng, WANG Jun, SHI Jun, et al. Review of artificial intelligence techniques in imaging data acquisition, segmentation, and diagnosis for COVID-19[J]. IEEE Reviews in Biomedical Engineering, 2021, 14: 4-15.
[9] YUE H, YU Q, LIU C, et al. Machine learning-based CT radiomics method for predicting hospital stay in patients with pneumonia associated with SARS-CoV-2 infection: A multicenter study[J]. Annals of Translational Medicine, 2020, 8(14): 859.
[10] GAO Kai, SU Jianpo, JIANG Zhongbiao, et al. Dual-branch combination network (DCN): Towards accurate diagnosis and lesion segmentation of COVID-19 using CT images[J]. Medical Image Analysis, 2021, 67: 101836.
[11] OULEFKI A, AGAIAN S, TRONGTIRAKUL T, et al. Automatic COVID-19 lung infected region segmentation and measurement using CT-scans images[J]. Pattern Recognition, 2021, 114: 107747.
[12] ABDEL-BASSET M, CHANG V, HAWASH H, et al. FSS-2019-nCov: A deep learning architecture for semi-supervised few-shot segmentation of COVID-19 infection[J]. Knowledge-Based Systems, 2021, 212: 106647.
[13] ISENSEE F, PETERSEN J, KLEIN A, et al. Abstract: nnU-net: Self-adapting framework for U-net-based medical image segmentation[M]∥Informatik aktuell. Wiesbaden: Springer Fachmedien Wiesbaden, 2019: 22-22.
[14] AN P, XU S, HARMON SA, et al. CT Images in COVID-19 [EB/OL]. https:∥doi.org/10.7937/tcia.2020.gqry-nc81, 2020-12-10.
[15] DESAI S, BAGLAL A, WONGSURAWAT T, et al. Data from chest imaging with clinical and genomic correlates representing a rural COVID-19 positive population [EB/OL]. https:∥doi.org/10.7937/tcia.2020.py71-5978, 2020-12-17.
[16] ÇIÇEK Ö, ABDULKADIR A, LIENKAMP S S, et al. 3D U-net: Learning dense volumetric segmentation from sparse annotation[C]∥Medical Image Computing and Computer-Assisted Intervention-MICCAI 2016, 2016: 424- 432.
[17] MILLETARI F, NAVAB N, AHMADI S A. V-net: Fully convolutional neural networks for volumetric medical image segmentation[C]∥2016 Fourth International Conference on 3D Vision (3DV). Stanford, CA, USA. IEEE, 2016: 565-571.
[18] SELVARAJU R R, COGSWELL M, DAS A, et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization[J]. International Journal of Computer Vision, 2020, 128(2): 336-359.


