1 引言
LCI是近几年Fujifilm公司研发用于临床医用的改革创新型技术。该集团所推出的内窥镜系统采用多光源整合技术,同时具备短波光以及白光,使用激光窄带短波混合白光显像,与普通白光下的图像即WL(White Light)图像相比,LCI图像能够更好地区分色差,可以呈现出更为清晰精准的图像,提高病变组织的可识别性[1-3]。
LCI内镜技术主要帮助医生了解消化道病变情况,便于筛选出早期胃癌、消化道肿瘤等疾病。目前LCI图像仅依靠医生肉眼观察进行判断,工作负荷较重,容易出现部分病变区域被忽略的情况,从而影响诊断结果。因此针对LCI图像的病变区域智能检测方法成为一种十分重要的临床辅助技术,然而目前对于LCI方面的图像研究论文较少,作为“智能”重要组成部分的深度学习在LCI图像上的研究更是凤毛麟角,本文主要将深度学习应用于LCI图像,实现LCI图像病变区域的智能检测。
深度学习是通过多种线性与非线性结构层组成的计算模型,分层提取特征,对数据进行高层抽象,尝试从数据中学习高层特征,学习数据之间的分布式特征表示[4- 6]。神经网络是深度学习的一种学习架构,可以看成是从点到线再到面的过程。“点”指的是神经网络中的计算节点,由这些节点组成一层,表示为对数据的某个抽象表达式, 节点的不同连接方式形成不同的处理层,这些处理层构建起来形成神经网络。
卷积神经网络是一种前馈神经网络,应用十分广泛,如目标检测[7]、人脸识别[8]等方面。其主要由卷积层、激活函数、池化层以及损失函数构成。卷积层的作用类似于滤波器,与输入相连的卷积层用来检测低阶特征,比如线条、角等,再到下一个卷积层,检测目标成了低级特征的组合,比如线条和角组成的矩形等。随着卷积层数的增加,需检测的特征越来越复杂,包含的语义信息(包括但不仅限于图像、语音等形式所包含的内容信息)也越来越丰富[9],但是卷积层的操作是一系列线性变换的函数,若整个网络都是线性,则模型对数据的表达能力不够强,因此需要在卷积层之间穿插激活函数,引入非线性成分,使得模型对于高维数据具有更好的学习能力。池化层降低了模型参数冗余度,减少计算量[10]。损失函数是用来衡量模型好坏的标准,通过损失函数能够对模型进行后续的改善与优化。
本文主要研究LCI图像中病变区域的智能检测,使用一种端到端的全卷积网络方法,选择适用于LCI图像病变区域与非病变区域界限模糊问题的损失函数,训练网络模型,实现对LCI图像的逐像素分类确定病变区域与非病变区域边界,从而确定病变区域范围,减少医生工作量,辅助医生进行病情诊断。将改进后的算法与传统的语义分割算法GrabCut[11]相比,检测的病变区域范围更加准确细致。
本文贡献如下:(1)将深度学习方法应用到医学领域的消化道内镜检查;(2)针对LCI图像在病变与非病变区域的分界线模糊问题,在FCN[12]的基础上重新设置损失函数;(3)通过实验验证了改进后的FCN算法检测效果较好,准确率达到94%。
本文安排如下:第2节介绍LCI图像的相关应用背景以及两种语义分割算法;第3节详细叙述本文所针对的问题以及改进算法;第4节展示本文改进算法与FCN以及GrabCut的对比结果;第5节为本文的结论。
2 LCI图像与语义分割算法
2.1 LCI图像介绍
LCI技术是一种新型内窥镜技术,在传统的内窥镜系统中加入激光光源,捕获到的图像在经过独特的图像处理技术之后,颜色对比更加明显,病变区域与周围粘膜之间的色彩差异更大,使病症更容易被判别[13-14],如图1是LCI图像与传统内窥镜下白光图像(WL图像)的对比,从图中我们可以看出LCI可以提供更清晰精准的图像。
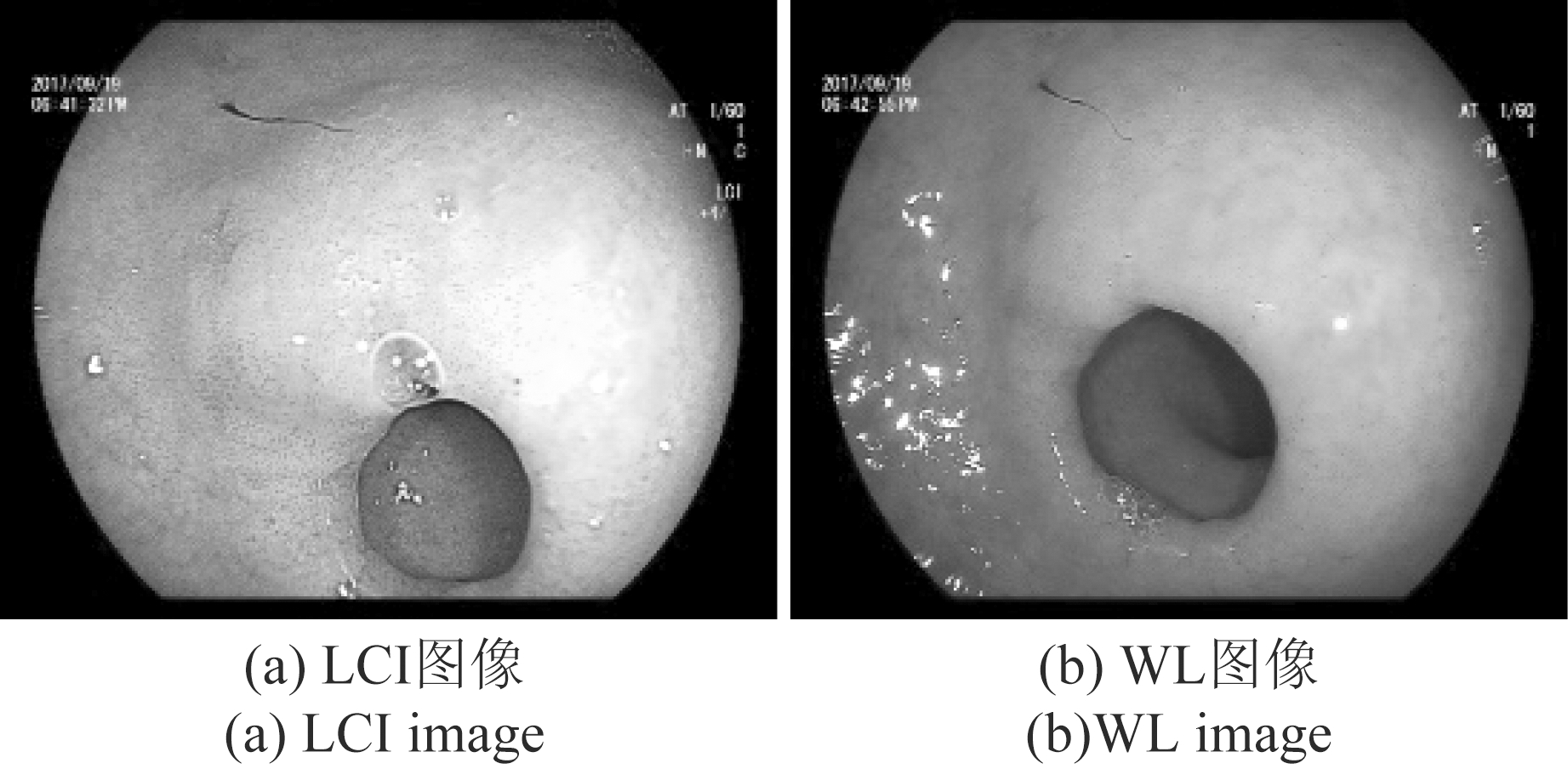
图1 LCI与WL图像对比
Fig.1 Comparison of LCI and WL
LCI技术常应用在上消化道与下消化道的检查。在上消化道中,常用来检查胃炎与早期胃癌症状,在粘膜色彩被增强后更容易观察到胃炎所造成的胃粘膜弥漫性发红、粘膜萎缩等。在下消化道中,LCI常用来观察结肠粘膜颜色以及微血管形态从而检查溃疡性结肠炎、结直肠腺癌型息肉以及早期结直肠癌等病症[15]。
在病例检测中,炎症与肠化现象比较常见。图2(a)、2(b)、2(c)是部分病症在LCI下的显示图像,其中(a)在形状上有一定的凹陷面且存在较为明显的部分暗红色区域,属于低级别上皮瘤变并有一定炎症。(b)中存在紫红色区域,有一定程度的肠化现象。(c)中存在息肉,在息肉周围有部分暗红色区域,属于炎症现象。
如图2所示,在不同病症中,病变区域都存在不同程度上的泛红现象,且在形状上与非病变区域的差异也较为明显,如(a)(c)中病变区域分别存在着凹陷与凸起的现象,在(b)中病变区域不仅呈现出紫红色,其纹理也与周围非病变区域存在差异。
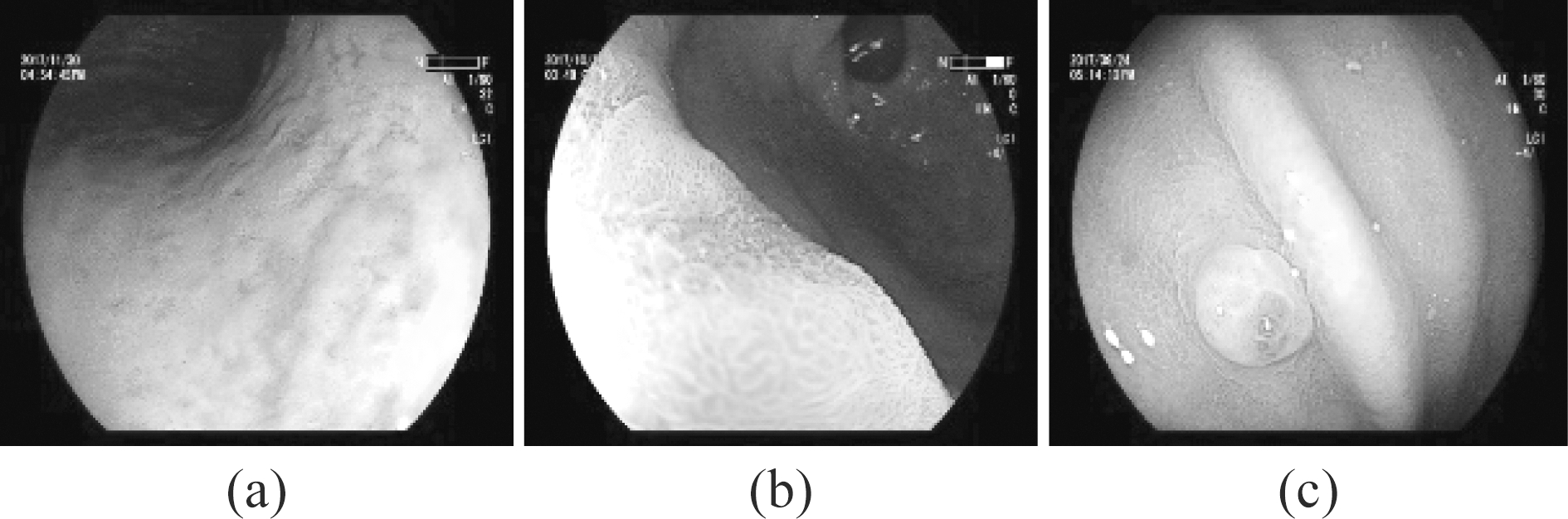
图2 LCI 病症图像
Fig.2 LCI illness images
2.2 GrabCut算法
GrabCut是传统的语义分割算法,根据颜色、形状等特征把图像划分成若干个互不相交的区域,分割图像中的不同物体,如前景与背景的分割。在GrabCut中,采用含有K个高斯分量的混合高斯模型分别对前景目标与背景进行建模。GrabCut算法需要少量的交互操作,由用户框选图像的前景目标,框选后的框外各个像素点均为图像背景像素,属于背景模型中的某一个高斯分量。框内则表示未知情况(可能为前景,也可能为背景),暂时将框内像素点全部归属于前景模型,估计前景与背景模型中高斯分量的参数。完成算法初始化后,需要对框内的未知像素点重新分配,将框内像素点代入到前景或背景模型中进行计算,更新两种模型中的像素集合以及模型参数,不断地进行迭代计算,直到模型达到稳定,参数不再更新为止即模型已经收敛。
GrabCut算法利用人为提供的背景像素集进行初始化,根据像素在前景模型与背景模型中的概率值以及像素之间的相似性对模型迭代更新直至模型收敛。若背景信息较为复杂或前景与背景差异小,该算法则难以准确分割,因此GrabCut仅能分割较为简单的图像,且每张图像都需要提供背景信息,进行迭代优化。
2.3 FCN算法
深度学习中的全卷积网络FCN是对图像进行像素级分类的语义分割算法。FCN是针对语义分割问题第一个实现端到端训练的算法,它在原有的分类网络(VGG[16],AlexNet[17]等经典网络模型)基础上进行微调。在分类网络中使用全连接层使网络输出为标量从而得到分类标签值,在语义分割中,我们需要得到图像的分割图,网络输出必须是二维数据,因此在FCN中将分类网络中的全连接层替换成卷积层。
在卷积网络中为了降低参数冗余度,减少计算量,使用了池化操作从而使得提取到的特征图尺寸变小。为了将卷积层生成的特征还原到像素空间中,得到与输入图像相同尺寸的分割结果图, FCN采用转置卷积方法[18]对网络输出结果进行上采样使其恢复到与输入图像相同的尺寸。若直接对最后一层的输出结果进行上采样得到的分割图会因为丢失许多低层特征的细节信息而显得比较粗糙,因此FCN中设计了跳跃性的网络结构来优化最终分割结果。跳跃性结构是将不同池化层的结果进行上采样再结合起来,使卷积层输出的高层粗粒度信息与低层特征信息都能够被利用到。使用Softmax函数定义损失函数,输出像素分类结果,映射在二维图像中,实现密集的像素输出预测。
结合不同池化层上采样的结果可以组合成不同的跳跃性结构,在FCN中有三种结构,分别为FCN-32s,FCN-16s及FCN- 8s。在FCN中已对这三种结构进行比较,其中FCN- 8s在PA(Pixel Accuracy,像素精度)、MPA(Mean Pixel Accuracy,均像素精度)以及MIOU(Mean Intersection over Union,均交并比)等指标均优于其他两种结构[12],因此本文在FCN- 8s的基础上改进与应用。
3 基于FCN算法的改进
本文研究目的是检测LCI图像中的病变区域,但目前LCI病症图像数量较少,训练时容易出现过拟合状态,难以保证训练出较好的网络模型。因此在训练网络之前,我们需要对LCI图像进行预处理,使用数据增强的方式人为增加训练图片数量。本文使用FCN中的跳跃性网络结构作为基本结构,更改损失函数,并重新设置网络参数来训练网络模型。
3.1 本文算法流程
本文主要工作是训练网络模型完成对LCI图像的逐像素分类,并通过对像素分类结果的处理得到LCI图像中病变区域与非病变区域的边界线,如图3为算法流程简图。由图3可知,本文以LCI图像作为网络输入,通过本文网络结构(在3.2节中详细介绍)实现对像素的分类,由于本文将LCI图像分为病变区域与非病变区域两部分,故像素的分类结果只有属于病变区域或属于非病变区域这两种情况,网络的输出是二值图像,图3中网络输出图的白色区域表示病变区域,像素值为1,黑色表示非病变区域,像素值为0,因此病变区域与非病变区域的边界区分可看作是寻找与像素值0相邻且像素值为1的像素点(边缘像素),因此若某一个像素点的像素值为1且以该像素点为中心与其相邻的八个像素点中存在像素值为0的情况,则认为该像素点为边缘像素,由此可提取出二值图像中病变区域(白色区域)的轮廓。将轮廓提取图与原图结合起来可得到本文最终结果,实现对LCI图像病变区域的检测。
3.2 网络结构
图3中所提到的“网络结构”是以跳跃性结构作为基本结构,跳跃性结构能将不同卷积层提取的特征结合在一起,如图4为本文所使用的网络结构模型,图中Conv、Relu以及Pool分别指的是卷积层、激活函数以及池化层。其中卷积层通过卷积核与输入数据进行线性计算提取图像特征,在卷积层后引入激活函数Relu,将输入的负值全部置为0,而正值不变,这种单侧抑制的做法能使模型将相关特征展现出来。池化层是对特征的降维,本文使用最大值池化,在特征图中每一个2*2的区域中选择一个最大值,即池化结果中每一个元素表示一个2*2子区域中的最大值,因此经过池化后图像尺寸变为原来的1/2,图像面积变为原来的1/4,池化操作能够大量的减少下一层的参数,加快运算速度。图4中的Drop层与池化层作用类似,但Drop层并不是取子区域中一个元素代表这个子区域,而是在模型训练时随机让网络某些节点不参与运算,网络权重不进行更新,从而达到加快运算速度,防止过拟合的目的。deconv是利用转置卷积进行的上采样操作,是跳跃性结构中的重要部分,能够将不同层的特征信息结合起来。

图3 算法流程简图
Fig.3 Flow chart of our algorithm
在该网络结构中,共使用了五层池化层。每次池化操作将图像尺寸缩小为原来的1/2,所以图像的最后输出尺寸为原来的1/32。为了将低层特征信息与高层信息结合起来,先使用转置卷积对最后一层的输出结果进行两倍上采样,使其与第四层池化层的输出结果维度一致,将两者进行融合后再进行两倍上采样后与第三层池化层的输出结果结合起来,最后将结果进行八倍上采样,使其与原始图像尺寸一致,再将其作为输出结果。
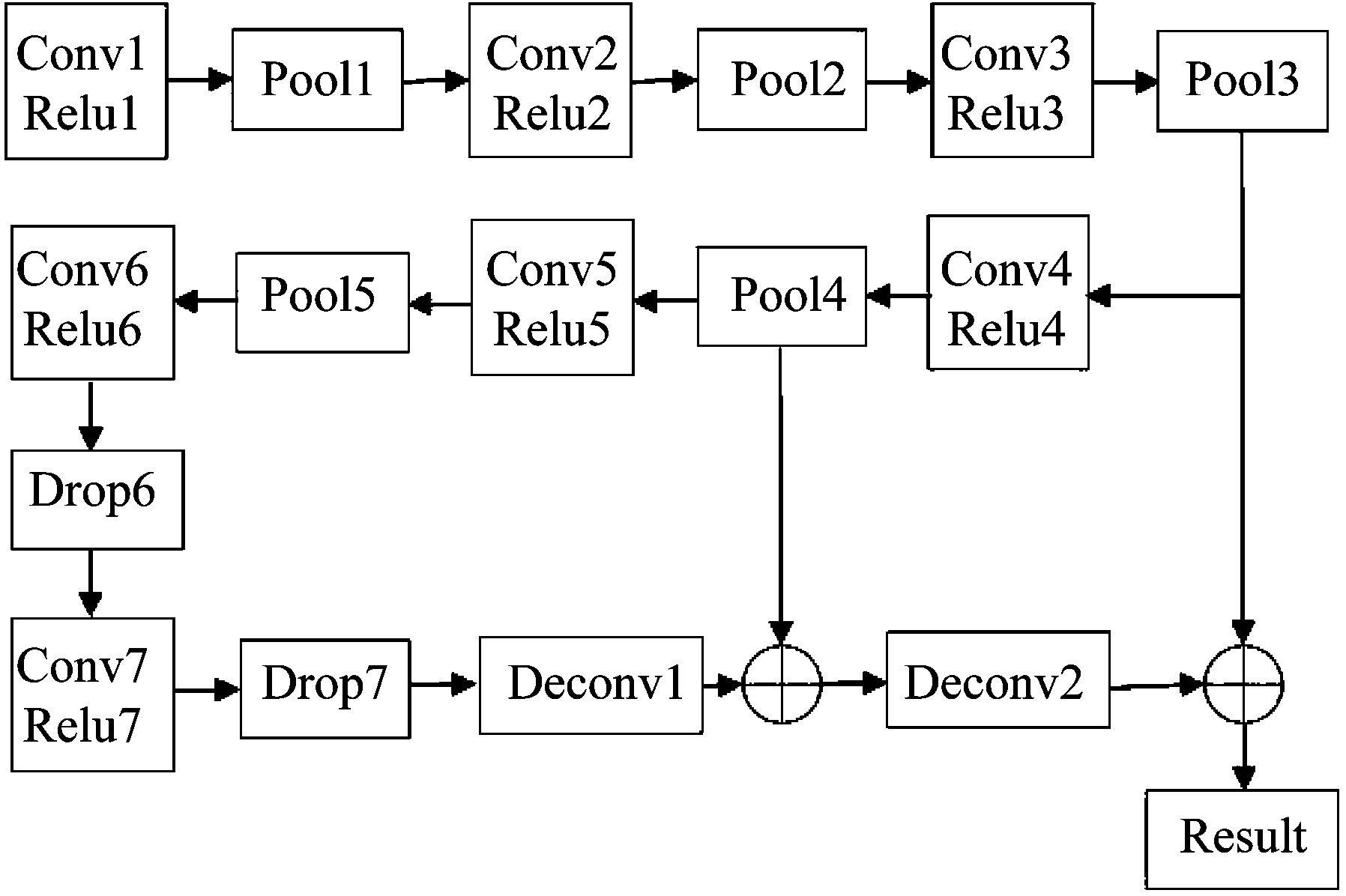
图4 网络结构
Fig.4 Network structure
3.3 损失函数
损失函数是用来计算模型预测值与真实值之间的差异程度,根据损失函数实现对模型参数的迭代更新,因此损失函数的选择对于模型的训练至关重要。FCN中使用Softmax判断分类结果概率分布,Softmax函数本质上是对数据做数学变换,将一个含任意实数的多维向量变换成范围在(0,1)之间的相同维数的向量,且变换后的向量所有元素之和为1。
由上文可知病变区域与非病变区域的区分可以作为二分类问题处理,将病变区域定为类别1,非病变区域定为类别2。对于单个像素点xi,其标注类别为y(y=1或y=2),在经过网络函数处理后,得到一个二维向量[sj, sy](j=1或j=2,j ≠ y),通过Softmax函数变换后可看作是xi属于类别1或者类别2的概率,使用交叉熵计算单个像素点的损失函数如式(1)所示,整个图像的损失函数等于各个像素点损失值之和,其中交叉熵表示用非真实概率分布(预测概率分布)模拟真实概率分布所需要的信息,可以衡量预测结果与真实结果之间的差异程度,两者差异越大,交叉熵越大。
(1)
在本文中用于网络训练的LCI图像中病变区域由医生划分,人为标注只能划分出病变区域大致范围,且不同医生在标注时会存在一定差异,如图5中两张图为不同医生标注情况,可以看出病变区域大体一致,但是很难在像素级别上做到精准标注。
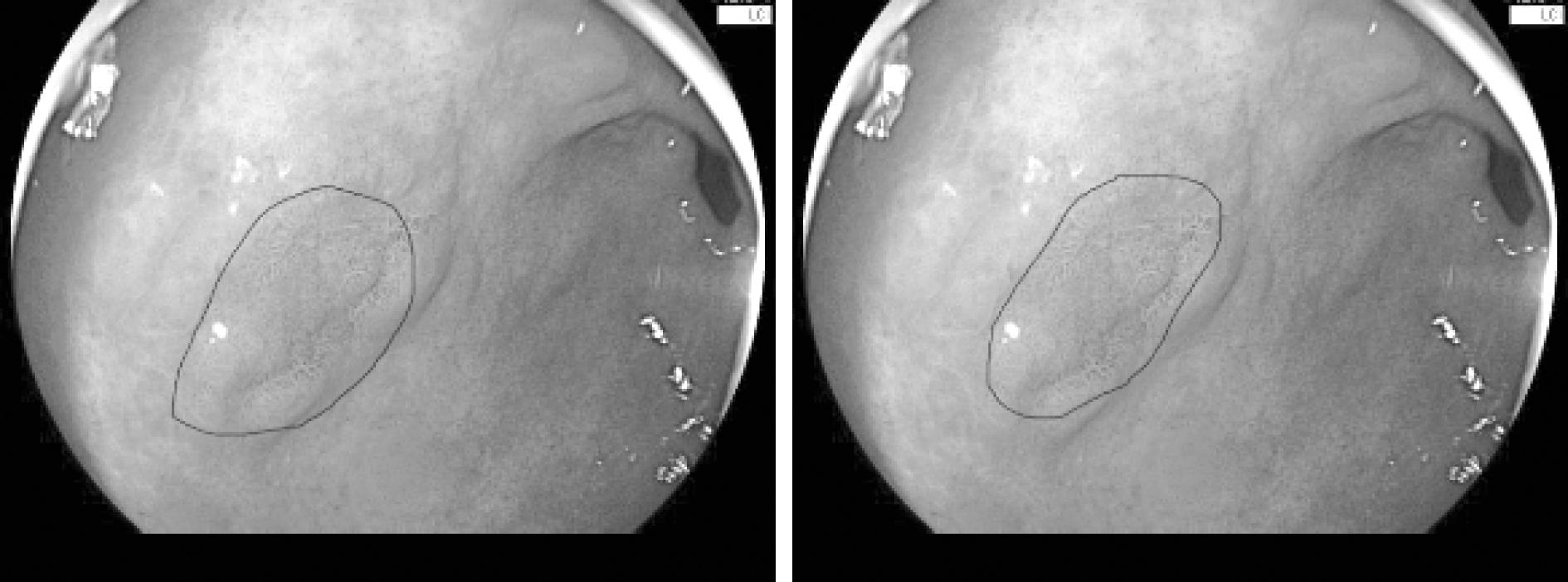
图5 图像标注
Fig.5 Images labeling
在训练时部分标签不准确会使部分非病变像素被当作病变像素进行处理,同时也存在将病变像素当作非病变像素处理的情况。若使用Softmax作为损失函数,其曲线如图6所示,当标注类别分数sy高于其他类别分数sj时,模型也会不断进行更新,若标注类别出现错误,根据Softmax的函数特性,这部分错误样本的作用范围会进一步扩大,影响模型性能。因此需要设置新的损失函数,降低错误样本的影响。SVM函数直接计算标注类别分值与其他类别之间的差距,设定间隔值。同样以单个像素点xi为例,如式(2)所示,γ是人为设定的参数值,它的意义在于确保标注类别分数sy必须比其他类别分数sj高γ才可认为这次分类合理,损失函数为0,SVM函数曲线图如图6所示。
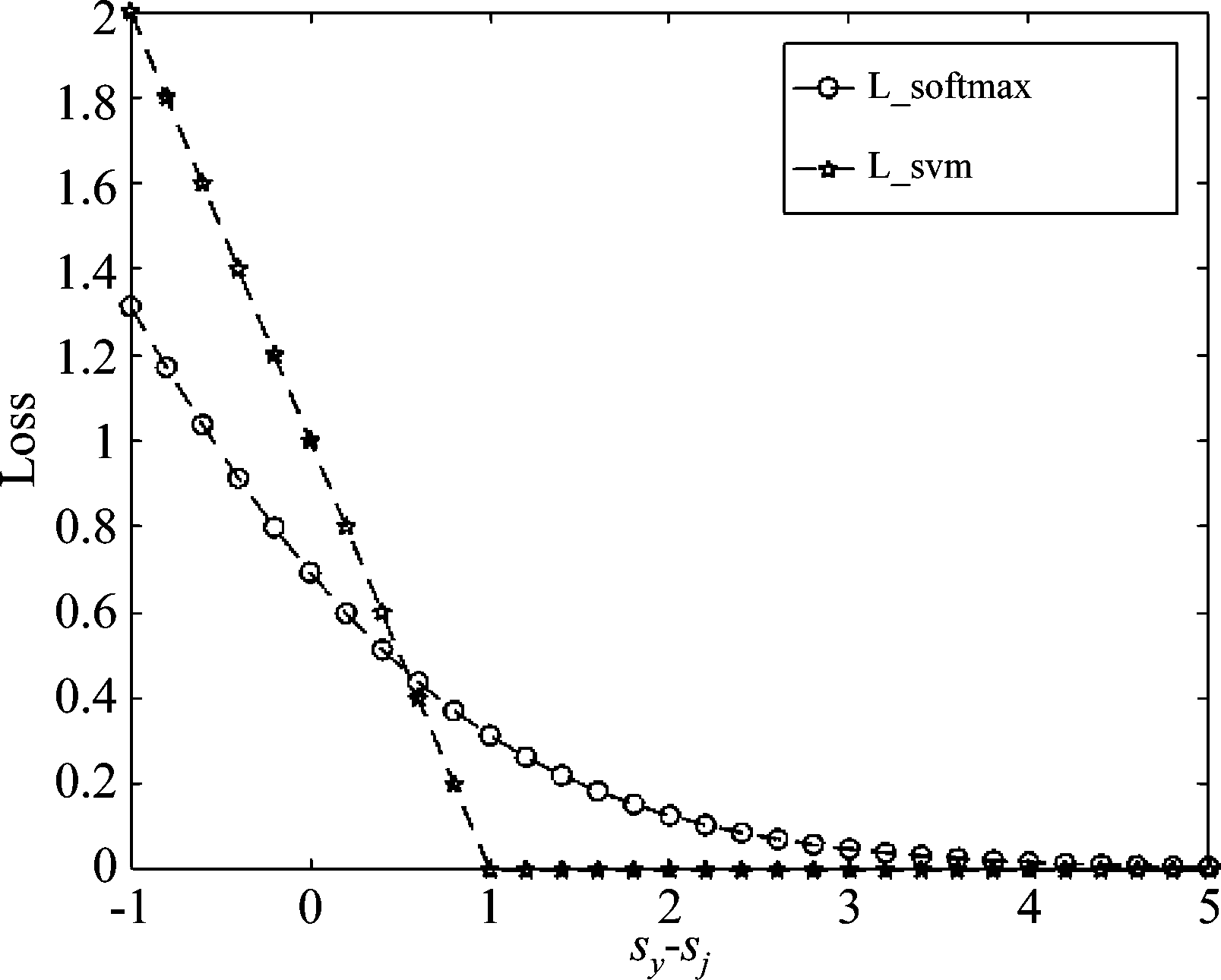
图6 损失函数比较
Fig.6 Loss function comparison
Lsvm(xi)=max(0,sj-sy+γ)=max(0,γ-(sy-sj)
(2)
两者都是为了增大正确类别的分类分值,将SVM函数表达式中的γ设为1,当标注类别分数比其他类别高出1时,损失函数为0,与网络参数无关,网络模型不进行更新,而Softmax会继续优化,使标注类别优势更加突出。因此从原理上表明SVM函数作为损失函数更适用于本文研究情况,后续在使用SVM与Softmax的实验比较中,为避免损失函数与支持向量机混淆,将SVM称为FCN-SVM,Softmax称为FCN-Softmax。
4 实验
本文的研究目的在于实现LCI图像病变区域的智能判别,将本文提出的FCN-SVM方法与FCN-Softmax以及传统的语义分割算法GrabCut进行实验对比。实验对象主要涉及中低分化癌、低级别上皮瘤变、炎症、肠化、息肉等症状图像,观察三种方法对于病变区域的检测效果。
4.1 数据处理
数据增强是解决数据集数量少以及防止过拟合的常用手段。对图像进行平移、翻转、高斯噪声扰动等等方式,生成许多与原图像在位置,形状以及尺寸上存在差异但是具有相同分类标签的图像,也可以理解为在图像数量有限的条件下,人为地制造更多的图像使网络模型能够提取更加丰富的特征信息,提高网络模型的泛化能力。本文主要使用了四种图像增强的方式,分别是角度旋转、尺寸缩放、颜色变换以及水平翻转,如图7所示。
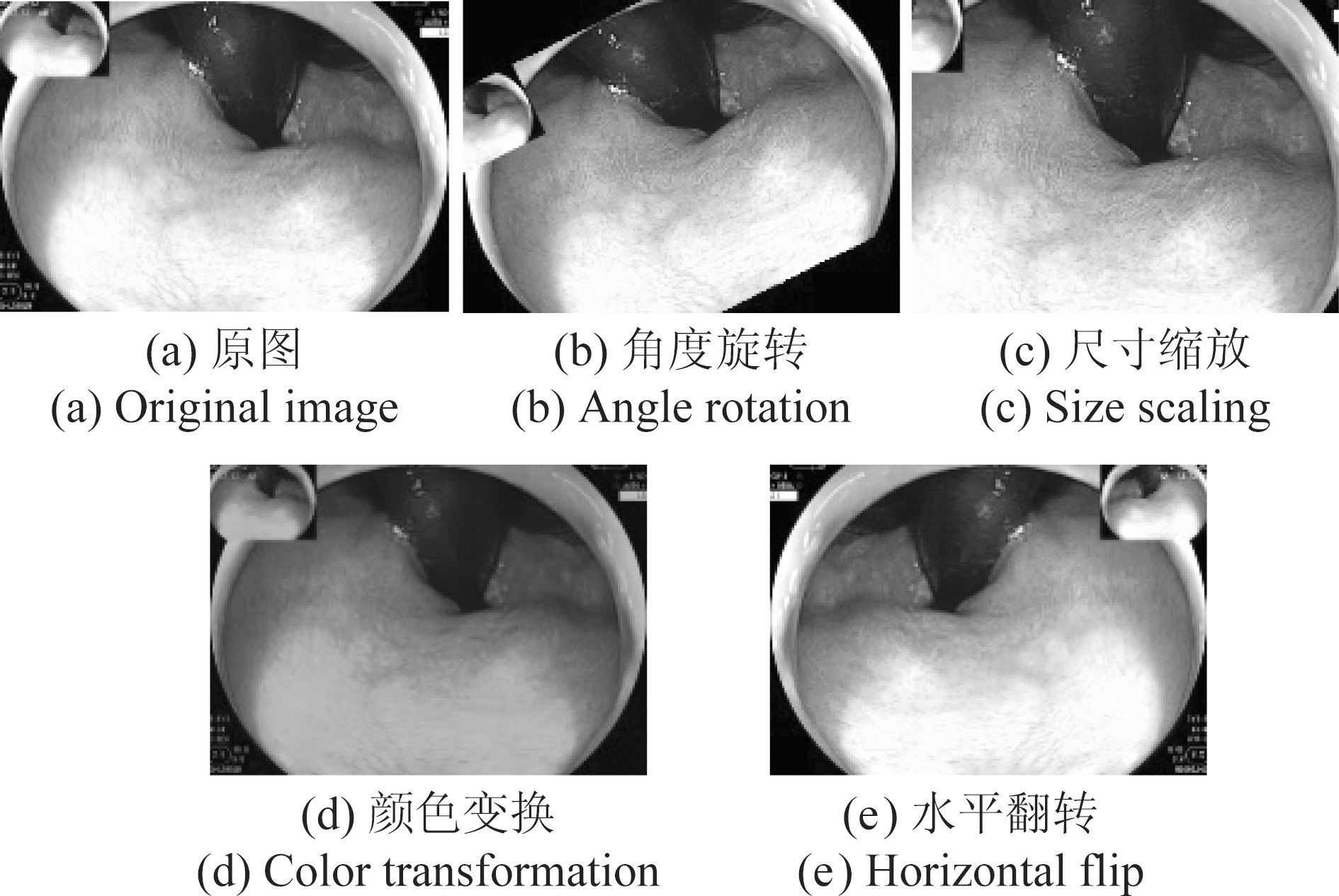
图7 数据预处理
Fig.7 Data preprocessing
其中(a)是原图,(b)、(c)、(d)、(e)为经过上述四种变换之后的图像,在经过数据增强处理后共有1200张图像。
4.2 实验与分析
本文实验在CPU为Intel i7-7820x,主频为3.60 GHz,内存为64G,显卡为GTX1080的电脑上进行。本文使用SGD[19](Stochastic Gradient Descent,随机梯度下降法)优化网络模型,设置初始学习率(决定网络权值更新速度)为1e- 8,权值衰减值(避免模型过拟合)为0.0005,使用上述参数对网络模型迭代优化,共迭代3000次,每1000次迭代将学习率降为原来的十分之一,完成训练过程。
本文主要将FCN-SVM与FCN-Softmax以及GrabCut进行实验对比。由第2.2节可以了解到GrabCut实质上是对框内的像素不断进行迭代更新计算,区分前景与背景。在实验中可将LCI图像中的病变区域当做前景,非病变区域当做背景,实现前景与背景的分割。在GrabCut中需要人为对图像设定初始框,界定背景元素。在实际应用中,为每张LCI图像手动定义背景信息以此来完成病变区域的检测是不合理的,因此本文设计固定尺寸为图像的初始框,本文所使用的实验图像尺寸均为720*576,为了让前景目标尽可能包含在目标框中,初始框目标尺寸不宜太小,本文选取初始框尺寸为550*550。本文共选取了六张不同形状且病变状况不相同的图像作为展示,如图8所示,白色框外确定为非病变区域,框内为待检测区域。
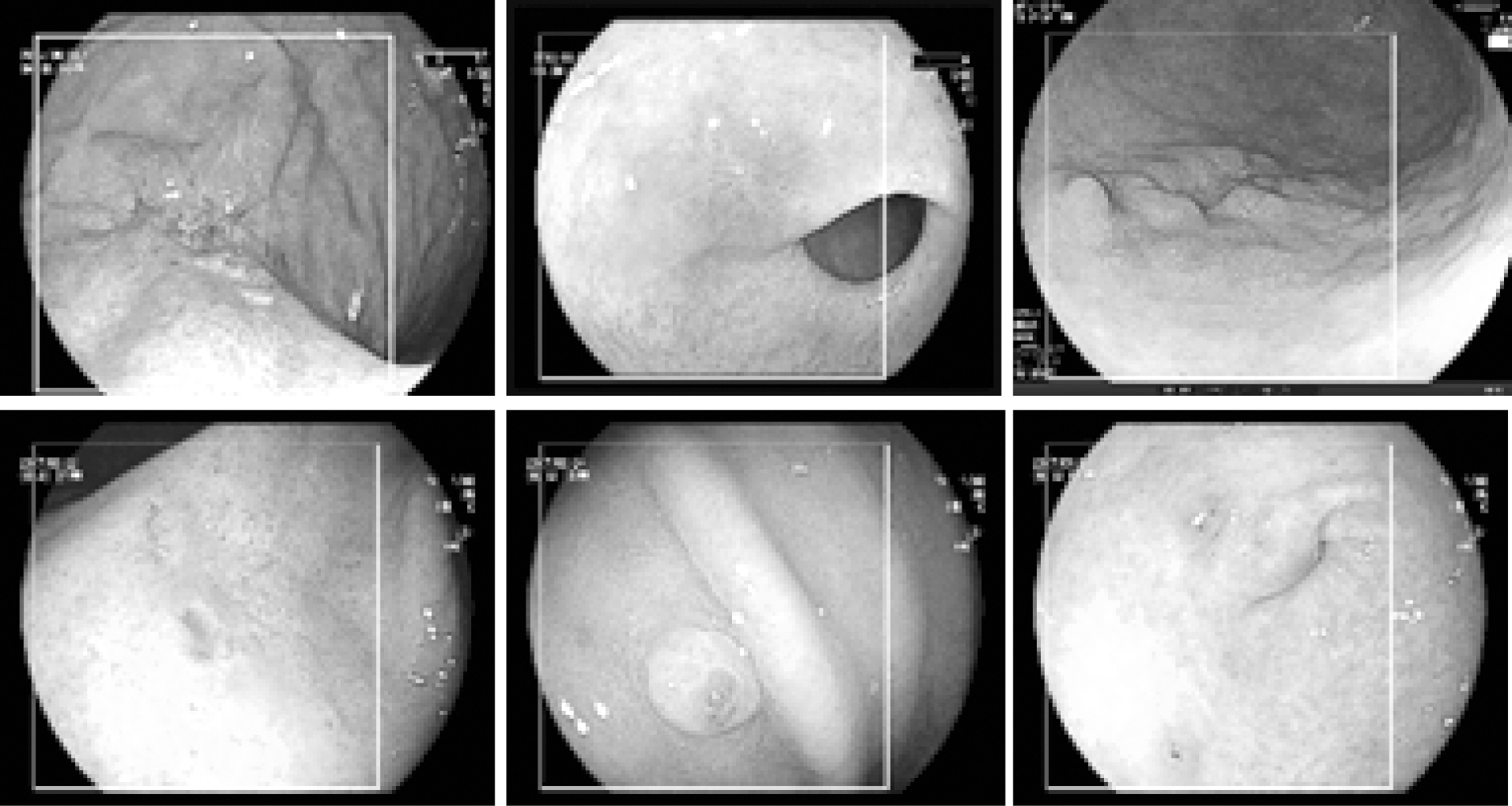
图8 GrabCut初始框标定
Fig.8 GrabCut initialization
在确定初始框位置之后,GrabCut开始对框内区域进行迭代计算,设置迭代次数N=20,50,100,如图9所示,迭代次数从50增加到100时,GrabCut分割结果不再发生变化,因此在N=50时,模型已经达到稳定,不再进行更新。
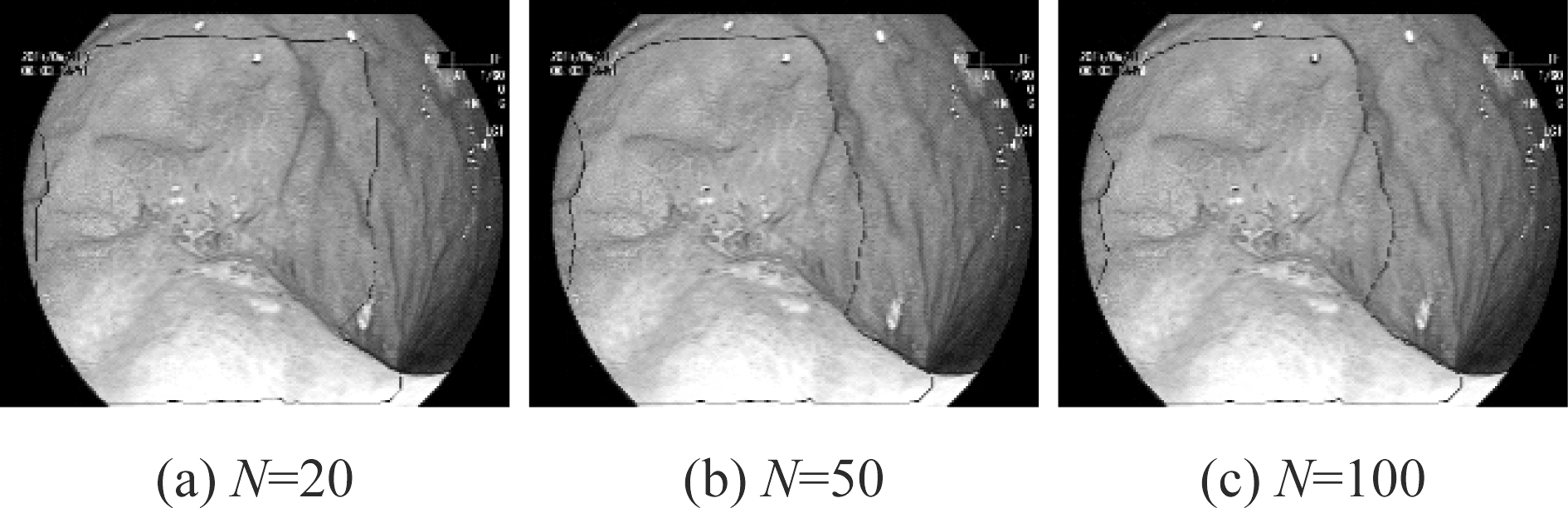
图9 N次迭代后的分割结果
Fig.9 Segmentation results after N iterations
FCN-SVM与FCN-Softmax以及GrabCut(迭代次数设置为50)的实验结果展示如图10第二、三、四行所示(绿色框内表示病变区域),图10中第一行Ground Truth作为实验结果对比参考标准,是经过两位医生共同标注处理后经多名专家审查完全符合病变区域的图像(黑色框内表示病变区域)。
从图10中可以看出,GrabCut对病变区域的判别区域普遍偏大,将许多非病变区域误检为病变区域,且对于息肉的检测十分不准确,无法判别息肉与正常褶皱。FCN-SVM与FCN-Softmax所检测到的区域基本都属于病变区域,但在FCN-Softmax方法下,不能完全覆盖到病变区域,例如不能将息肉完整判别出来,覆盖面较小,FCN-SVM则基本能够覆盖到病变区域,判别较为准确。
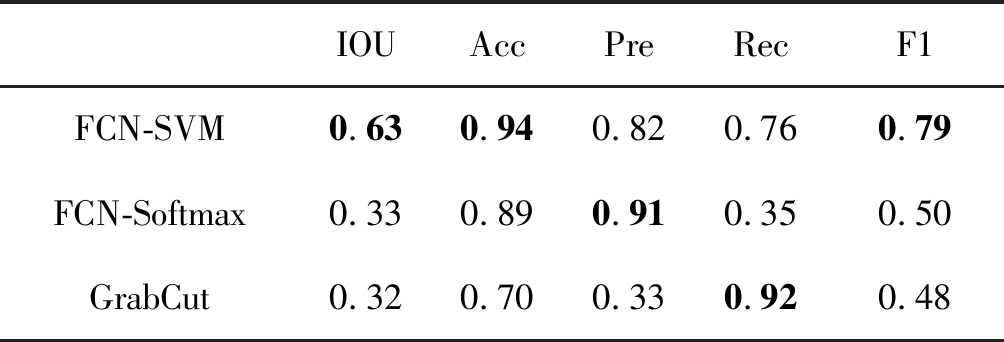
为了更严谨地判断检测效果,本文从交叠率(IOU)、准确率(Acc)、精确率(Pre)、召回率(Rec)以及调和平均数(F1)这五个方面对实验结果进行定量分析,具体数据如表1所示。
表1 模型评价指标对比
Tab.1 Evaluation comparisons
IOUAccPreRecF1FCN-SVM0.630.940.820.760.79FCN-Softmax0.330.890.910.350.50GrabCut0.320.700.330.920.48
表中IOU(Intersection over union)指检测结果与实际诊断区域的交并比,反映两者之间的重叠率,比值越高说明检测效果越好。准确率指对病变与非病变检测均正确的面积占整个图像面积的比例。精确率针对预测结果,表示在检测结果为病变的面积中真实病变面积所占的比例,精确率越高表明检测出的病变面积越精准。而召回率则针对原始数据,表示在真实病变区域中被检测到的病变面积所占的比例,召回率越高表明被检测出的实际病变面积越多。精确率与召回率同时越高说明检测效果越好,但是在某些情况下精确率越高,召回率就越低,反之亦然,因此使用精确率与召回率的调和平均数F1更能反映检测效果。
从表1及图10中可以看出,在三种算法中,FCN-Softmax精确率最高,但是召回率最低,检测到的病变区域基本符合真实病变区域,但检测面积较小,不能完全检测出真实病变区域。GrabCut则与之相反,召回率最高,精确率最低,检测到的病变区域比较全面,但检测面积过大,包含了许多非病变区域。而在FCN-SVM中,精确率与召回率数值则比较均衡,且在交叠率、准确率以及调和平均数上数值最高,检测到的病变区域比较全面且检测精准。

图10 FCN-SVM与FCN-Softmax、GrabCut对比实验
Fig.10 Comparison of FCN-SVM、FCN-Softmax and GrabCut
4.3 实验小结
本文实验主要展示FCN-SVM对病变区域的检测效果,从交叠率、准确率、精确率、召回率以及调和平均数这五个评价指标上与FCN-Softmax以及GrabCut进行对比,实验结果表明,FCN-SVM在交叠率、准确率以及调和平均数均优于FCN-Softmax以及GrabCut,在精确率和召回率这两种指标上比较均衡,检测到的病变区域比较全面,且检测精准。FCN-Softmax精确率虽高,但检测到的病变面积较小,对病变区域存在漏检的情况。而GrabCut检测出的病变面积过大,包含许多非病变区域,对病变区域存在误检的情况。FCN-SVM较少出现漏检以及误检情况,检测效果较为突出。
5 结论
本文首次将全卷积网络方法应用到消化道内镜检查的LCI图像中检测病变区域。在FCN的基础上,针对病变区域与非病变区域分界线模糊问题,使用FCN-SVM函数作为损失函数,重新设定参数,训练网络模型。实验结果表明FCN-SVM在交叠率、准确率以及调和平均数F1等指标上表现突出,检测出的病变区域更加全面准确,因此FCN-SVM在一定程度上能够减轻医生工作量,辅助医生对炎症、早期癌症等病情的诊断,加快诊断速度。
[1] 王菲, 陈石岩, 谭新华, 等. 联动成像内镜与白光内镜对胃黏膜感染幽门螺杆菌的诊断价值比较[J]. 实用医学杂志, 2017, 33(14): 2325-2328.
Wang Fei, Chen Shiyan, Tan Xinhua, et al. Diagnostic value of linked color imaging for Helicobacter Pylori infection compared with white light imaging[J]. The Journal of Practical Medicine, 2017, 33(14): 2325-2328.(in Chinese)
[2] Fukuda H, Miura Y, Hayashi Y, et al. Linked color imaging technology facilitates early detection of flat gastric cancers[J]. Clinical Journal of Gastroenterology, 2015, 8(6): 385-389.
[3] Min Min, Deng Pei, Zhang Wenhua, et al. Comparison of linked color imaging and white-light colonoscopy for detection of colorectal polyps: a multicenter, randomized, crossover trial[J]. Gastrointestinal Endoscopy, 2017, 86(4): 724-730.
[4] Deng Li, Yu Dong. Deep learning: methods and applications[J]. Foundations and Trends® in Signal Processing, 2014, 7(3- 4): 197-387.
[5] Arel I, Rose DC, Karnowski TP. Deep machine learning-a new frontier in artificial intelligence research[J]. IEEE Computational Intelligence Magazine, 2010, 5(4): 13-18.
[6] 李彦冬, 郝宗波, 雷航. 卷积神经网络研究综述[J]. 计算机应用, 2016, 36(9): 2508-2515.
Li Yandong, Hao Zongbo, Lei Hang. Survey of convolutional neural network[J]. Journal of Computer Applications, 2016, 36(9): 2508-2515.(in Chinese)
[7] 楼奇哲, 刘乐, 姚元. 采用卷积神经网络的海面多目标检测研究[J]. 信号处理, 2018, 34(9): 1053-1059.
Lou Qizhe, Liu Le, Yao Yuan. Multi-target detection in sea clutter with convolution neural network[J]. Journal of Signal Processing, 2018, 34(9): 1053-1059.(in Chinese)
[8] Schroff F, Kalenichenko D, Philbin J. FaceNet: A unified embedding for face recognition and clustering[C]∥Proceedings of The IEEE Conference on Computer Vision and Pattern Recognition, 2015: 815- 823.
[9] Zeiler M D, Frgus R. Visualizing and understanding convolutional networks[C]∥European Conference on Computer Vision. Springer, Cham, 2014: 818- 833.
[10] Schmidhuber J. Deep learning in neural networks: An overview[J]. Neural Networks, 2015, 61(61): 85-117.
[11] Rother C, Kolmogorov V, Blake A. Grabcut: Interactive foreground extraction using iterated graph cuts[C]∥ACM Transactions on Graphics(TOG). ACM, 2004, 23(3): 309-314.
[12] Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation[C]∥Proceedings of The IEEE Conference on Computer Vision and Pattern Recognition, 2015: 3431-3440.
[13] Kanzaki H, Takenaka R, Kawahara Y, et al. Linked color imaging(LCI), a novel image-enhanced endoscopy technology, emphasizes the color of early gastric cancer[J]. Endoscopy International Open, 2017, 5(10): E1005-E1013.
[14] Sun Xiaotian, Dong Tenghui, Bi Yiliang, et al. Linked color imaging application for improving the endoscopic diagnosis accuracy: a pilot study[J]. Scientific Reports, 2016, 6(33473): 1-7.
[15] 谢聪颖, 刘岩. 联动成像内镜的原理及临床应用现状[J]. 中华消化内镜杂志, 2017, 34(8): 600- 602.
Xie Congying, Liu Yan. The principle and clinical application of linked color imaging endoscopy[J]. Chinese Journal of Digestive Endoscopy, 2017, 34(8): 600- 602.(in Chinese)
[16] Simonyan K, Zisserman A. Very Deep Convolutional Networks for Large-Scale Image Recognition[C]∥International Conference of Learning Representation, 2015.
[17] Krizhevsky A, Sutskever I, Hinton G E. Imagenet classification with deep convolutional neural networks[C]∥Advances in Neural Information Processing Systems. 2012: 1097-1105.
[18] Zeiler M D, Taylor G W, Fergus R. Adaptive Deconvolutional Networks for Mid and High Level Feature Learning[C]∥International Conference on Computer Vision. IEEE Computer Society, 2011: 2018-2025.
[19] Chong E K P, Zak S H. An introduction to optimization[M]. John Wiley & Sons, 2013.


