1 引言
恶性脑肿瘤是世界上最可怕的癌症类型之一,通常会使患者的认知功能下降、生活质量变差。成人中最常见的脑肿瘤是原发性中枢神经系统淋巴瘤和胶质瘤,其中胶质瘤占到恶性肿瘤的80%以上[1],因此胶质瘤是肿瘤分割的重点对象。但由于胶质瘤可出现在大脑的任何位置且大小不定、形状不规则,使得其分割仍然是一个具有挑战性的任务,因此如何采用现代信息技术高效且全自动化地分割脑肿瘤成为一个重要的研究方向[2]。核磁共振成像(Magnetic Resonance Imaging, MRI)技术具有非侵入性,对脑肿瘤图像分割具有重要的辅助作用[3- 4]。脑肿瘤MRI序列包括T1加权(T1-weighted)、T1C(Contrast enhanced T1-weighted images)、T2加权(T2-weighted images)及FLAIR(Fluid Attenuated Inversion Recovery)等成像序列,临床上通常结合四种图像共同诊断肿瘤的位置和大小。
近年来研究人员运用多种方法来分割脑肿瘤图像,总体上可分为无监督学习算法[5-7]和监督学习算法[8-12]。无监督学习算法大都属于半自动算法且分割精度总体不高,Levine等[5]提出的区域生长法需要在分割图像中手动设置初始点;Szilagyi等[6]提出将多个模糊C均值(Fuzzy C-means, FCM)算法级联的模型,但此算法仅在有限数据集上进行了测试,模型泛化程度并不高;童云飞等[7]采用快速模糊C均值选择肿瘤区域内的初始轮廓,而后利用水平集方法来提取脑肿瘤边界,但初始轮廓选择不正确会导致分割结果不理想。
对比上述无监督学习算法,监督学习算法由于标签的反馈机制使得算法整体分割精度变高。与极端随机树(Extremely Randomized Trees, ERT)[8]和支持向量机(Support Vector Machine, SVM)[9]等传统监督机器学习算法相比,基于卷积神经网络(Convolutional Neural Network, CNN)的方法不依赖于人工提取特征,而是自动从训练数据中学习到高水平和任务自适应的复杂特征,使得算法的精度越来越高。Havaei等[10]提出一种级联的CNN模型来提高脑肿瘤的分割精度,但网络模型复杂度过高导致训练较困难,分割精度(文中无特指情况下,分割精度代表相似系数Dice指标)只能达到85%左右;Pereira等[11]为了加快网络的训练速度,在CNN模型中用多个3×3小尺寸的卷积核来减少网络参数同时加深层数提高模型精度,但分割精度依然没有超过90%;师冬丽等[12]结合模糊推理系统,建立学习规则对CNN预测肿瘤点的概率进行再判断虽然分割精度可达90%,但算法却变为半自动化。目前基于CNN的脑肿瘤图像分割精度难以提升主要由于:(1)相邻像素块的标签具有独立性,没有考虑标签之间的相关性;(2)像素块的大小限制了感受野的大小,网络只能提取到局部特征,导致CNN无法分割出精细的肿瘤边界,精细即与脑肿瘤图像标签相比,算法可正确分割出脑肿瘤的总体轮廓,精度可达85%以上且不存在过多分割散点,分割出的脑肿瘤边界更加突出;(3)CNN针对每个相邻像素块逐个卷积,计算复杂度较高。
针对CNN分割图像的缺陷,文献[13]提出的马尔科夫随机场算法和文献[14]、文献[15]提出的条件随机场算法考虑到图像标签之间的相关性,将其作为CNN的后处理手段用于图像的边界分割;文献[18]提出一种不依懒于像素块直接对像素点分割的全卷积神经网络,大大降低了计算复杂度。基于此本文提出一种基于改进的全卷积神经网络(Fully Convolutional Neural Network, FCNN)和条件随机场(Conditional Random Fields, CRF)的全自动脑肿瘤MR图像分割算法。首先对多模态MR脑肿瘤图像做灰度归一化及灰度图像融合的预处理,而后对预处理图像建立FCNN模型进行粗分割,随后基于图像标签之间的相关性,将概率图谱模型CRF融合到FCNN中,进行精细的边界分割,提高脑肿瘤的分割精度。
2 基本理论
2.1 改进的全卷积神经网络
1998年LeCun等[16]将CNN首次应用于图像识别领域,2012年由Krizhevsky[17]等提出的AlexNet网络结构在CNN物体分类领域内取得重大突破,2015年Long等[18]提出了一种像素级别的图像语义分割网络—全卷积神经网络FCNN,该网络在AlexNet基础上改进,可端对端地对每个像素分类,实现图像分割任务。FCNN将AlexNet的两个全连接层转化为卷积层使得网络最后的输出仍为二维矩阵,这样保留了像素间的空间信息,更加有利于特征的提取。为使网络的输出和输入大小一致,原始FCNN直接对最后一个卷积层的输出进行上采样,使其恢复到输入图像相同的尺寸,但这种操作只利用了第5次池化的特征信息,得到的目标特征较粗糙,难以实现目标的精确分割。
本算法将利用不同池化层的特征信息,实现特征信息的多级融合。在对最后一个卷积层的输出进行上采样后,首先与第四次池化的特征向量矩阵进行第一次融合;而后对得到的特征融合矩阵进行上采样并与第三次池化的特征向量矩阵进行第二次融合;然后再将此融合特征矩阵进行上采样与第二次池化的特征向量矩阵进行第三次融合,最后将第三次融合的特征矩阵进行上采样得到与原始图像相同大小的特征矩阵,从而得到更好的特征信息矩阵。同时算法在每个卷积层后增加Ioffe等[19]提出的批量正则化(Batch Normalization, BN)层以加快网络的训练速度,提高模型的分割精度。多级特征信息融合的网络结构如图1所示,其中Conv为卷积层,该层可提取图像的边缘、位置等多种特征;Pool为池化层,可实现特征降维,保留卷积层提取的主要特征;BN为批量正则化层,可保证卷积后网络的权重分布不发生很大的变化;Up为上采样层,主要以反卷积的方式增大特征矩阵;Crop_Fuse对特征矩阵进行裁剪和融合操作,Pixelwise Prediction为像素分类预测层,通过对每个像素点分类实现图像分割。
2.2 条件随机场
基于CRF模型的图像语义分割是经典的概率图谱分割算法之一,可去除分割结果中的噪声,强化图像的边界分割。本算法将Zheng等[15]提出的全连接CRF模型融合到改进的FCNN网络中,进行脑肿瘤边界分割的后处理,提高脑肿瘤图像的分割精度。
在全连接CRF模型中,像素归属为所属标签的能量函数可表示为:
(1)
式(1)中:E(x)为像素归属标签的总能量;φu(xi)为一元能量势函数,表示不考虑像素与像素间的关系时,将像素i归属为标签xi的能量;φp(xi,xj)为点对能量势函数,表示基于像素间的灰度值差异和空间位置距离,将像素i、 j同时分割为标签xi、xj的能量,描述两两像素点间的关系,使得相似像素点分配相同的标签。
一元能量势函数φu(xi)可以从FCNN的初始粗分割结果中得到,点对能量势函数φp(xi,xj)可表示为:
(2)
式(2)中: μ(xi,xj)是标签兼容性矩阵,表示不同像素归属于不同标签的惩罚项,当xi≠xj时,惩罚项![]() 是高斯滤波器核, fi, fj为滤波器的特征向量,由像素间的空间位置及像素的灰度值来决定,m为滤波器的数量;
是高斯滤波器核, fi, fj为滤波器的特征向量,由像素间的空间位置及像素的灰度值来决定,m为滤波器的数量;![]() 为每一个滤波器的权重。
为每一个滤波器的权重。
在CRF模型中,采用双边高斯滤波器将空间上离得近并且灰度值接近的像素点划分为同一标签;利用空间平滑高斯滤波器去除脑肿瘤分割结果中孤立的小区域。所以式(2)可进一步表示为:
(3)
式(3)中:Ri和Rj表示像素i与j的灰度特征值向量;Pi和Pj表示像素i与j在空间中的位置关系特征向量;θα、θβ及θγ表示像素的灰度、位置等对势函数的影响权重。Zheng等[18]经大量实验表明,当双边滤波器权重![]() 控制参数θα=160、θβ=3,平滑滤波器权重
控制参数θα=160、θβ=3,平滑滤波器权重![]() 控制参数θγ=5时可较好地分割图像的边界。
控制参数θγ=5时可较好地分割图像的边界。
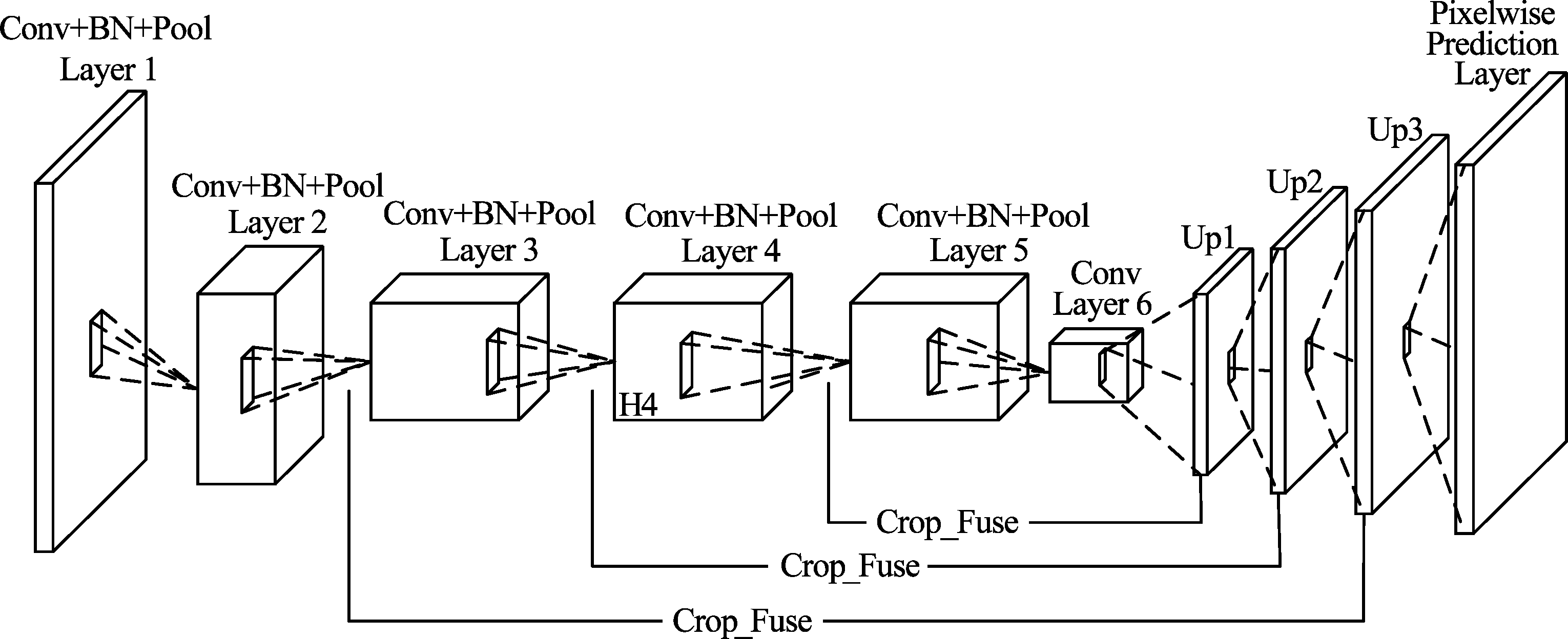
图1 多级特征信息融合的网络结构图
Fig.1 The network structure chart of the multilevel features information fusion
3 本文算法原理
本文改进的全卷积神经网络的脑肿瘤图像分割算法流程如图2所示。首先对归一化的多模态MR脑肿瘤图像数据进行灰度图像融合;随后将融合的部分数据作为训练集,部分数据作为测试集,利用训练集训练FCNN和CRF的融合算法;最后在测试集上进行模型的测试和分割结果的指标评价。
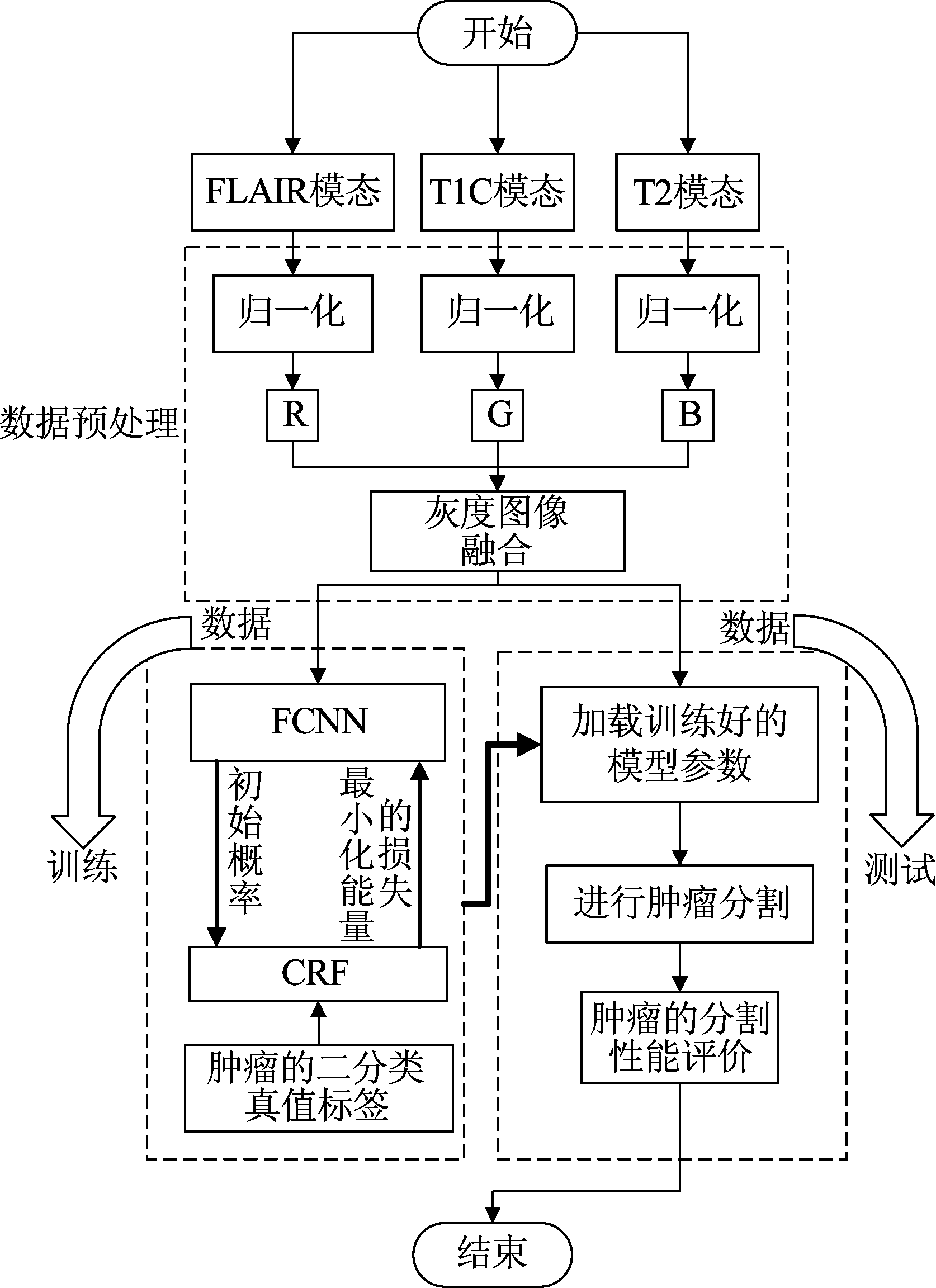
图2 算法流程图
Fig.2 The flow chart of algorithm
3.1 图像预处理
原始MR脑肿瘤图像中的每一个像素点在计算机中以16 bit进行存储,但在数字图像处理技术中都是对8 bit图像进行处理,故本文先对MR图像进行灰度归一化,将每一像素点的灰度值均匀压缩至范围0~255之间。考虑到神经网络只通过单一模态图像数据提取到的特征不足以精确地分割脑肿瘤边界,所以根据MR四种模态图像的成像特点,对FLAIR、T2和T1C三种模态分别作为R、G、B通道进行简单的灰度图像融合以便卷积核能学习到不同模态的不同特征。
3.2 改进的FCNN- 4s粗分割算法
为了获得良好的肿瘤特征,便于概率图谱CRF模型进行精细的边界分割。本文对原始的FCNN算法进行改进,提出精细的特征融合模型,同时为了加快网络的收敛速度,提高模型的精度,将BN层加入到网络中,形成图3所示的FCNN- 4s算法。其中Data为数据层,Conv为卷积层,Pool为池化层,ReLU(Rectified Linear Units)为激活函数层,Up为上采样层,Prob为输出的概率图谱层。Fuse为特征融合操作,融合在高维特征矩阵和低维特征矩阵的维数和大小相同前提下,将两目标矩阵对应维度的数据进行简单的相加运算。Crop表示如果融合前两目标矩阵的维数和大小不一致时(如表1中Pool4层输出大小为28×28×512,而Up1层输出大小为18×18×2),算法为使两目标矩阵维数和大小一致而进行的矩阵裁剪操作。当维数不一致时,算法将低维特征矩阵同1×1×Nhigh(Nhigh为高维特征矩阵数量)的卷积核进行卷积,使得两矩阵维数相同的同时又不会改变低维特征矩阵值的大小;当大小不一致时,因为图像的特征信息主要集中于中间部分,算法从低维特征矩阵的中心向两边对称提取与高维特征矩阵相同的大小,降低脑肿瘤特征信息的损失,使得其对分割结果几乎不产生影响。
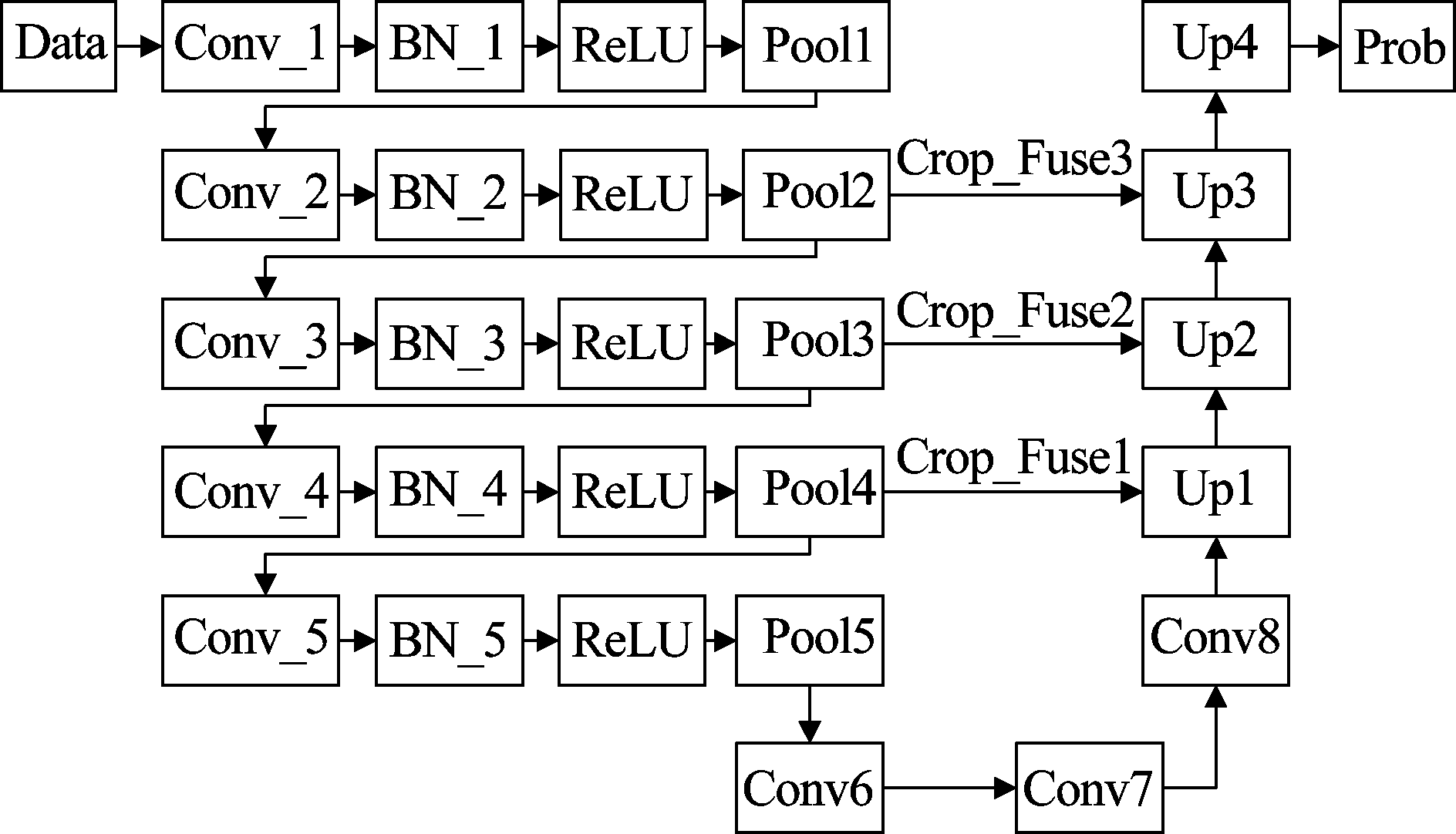
图3 改进的FCNN- 4s的网络结构图
Fig.3 The network structure chart of the improved FCNN- 4s
表1为图3网络的具体参数,其中Layer为每一层的名字;Kernel表示卷积核或池化核的维度和大小;Stride表示卷积运算过程中滑动窗口的步长;Pad表示计算前对输入矩阵进行一定的边缘扩充,为了防止池化后的特征信息矩阵过小,本文对原始脑肿瘤图像(240×240)进行Pad为100的操作;Output-Size表示输出特征矩阵的维度和大小。算法将不同维度上的特征矩阵进行三次融合,使获取的脑肿瘤特征更加精细,同时BN层可使每次迭代训练的参数权重分布不发生很大的变化以加快收敛速度。
表1 改进的FCNN- 4s的网络结构参数
Tab.1 The network structure parameters of the improved FCNN- 4s
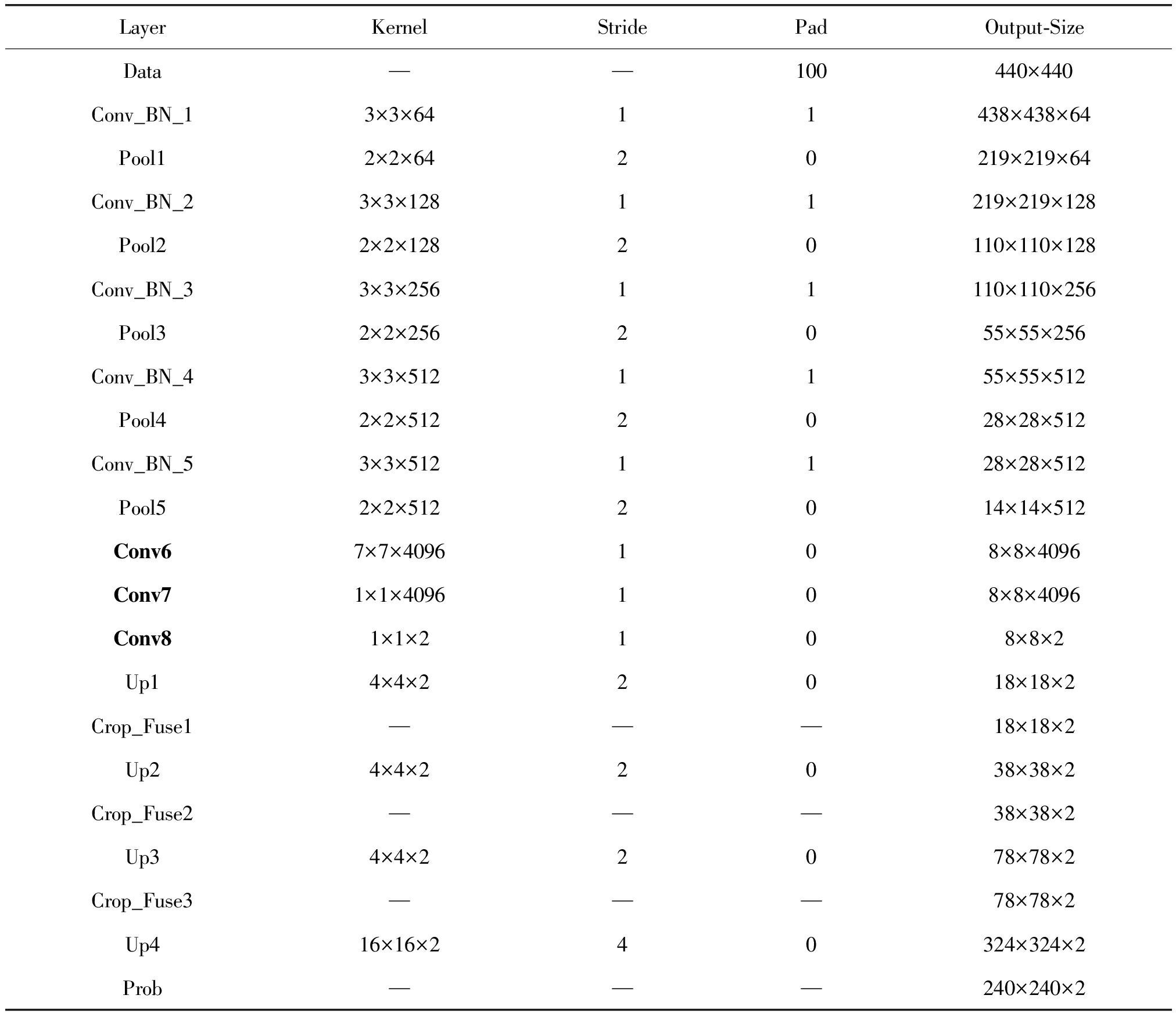
LayerKernelStridePadOutput-SizeData——100440×440Conv_BN_13×3×6411438×438×64Pool12×2×6420219×219×64Conv_BN_23×3×12811219×219×128Pool22×2×12820110×110×128Conv_BN_33×3×25611110×110×256Pool32×2×2562055×55×256Conv_BN_43×3×5121155×55×512Pool42×2×5122028×28×512Conv_BN_53×3×5121128×28×512Pool52×2×5122014×14×512Conv67×7×4096108×8×4096Conv71×1×4096108×8×4096Conv81×1×2108×8×2Up14×4×22018×18×2Crop_Fuse1———18×18×2Up24×4×22038×38×2Crop_Fuse2———38×38×2Up34×4×22078×78×2Crop_Fuse3———78×78×2Up416×16×240324×324×2Prob———240×240×2
3.3 FCNN- 4s和CRF的细分割融合算法
本文判断MR脑肿瘤图像中每一个像素点是否为肿瘤点,标签l中用0表示非肿瘤像素点,1表示肿瘤像素点,属于二分类问题。当获取到FCNN- 4s的两张初始概率图谱后,首先初始化能量函数得到像素点原始归属标签概率值,随后采用以下步骤计算CRF模型,不断迭代修正FCNN- 4s预测的两类概率图谱,融合算法如下所示。
步骤1 用高斯滤波器![]() 分别对l=0和l=1的两个类别概率图谱进行滤波,得到两类滤波结果
分别对l=0和l=1的两个类别概率图谱进行滤波,得到两类滤波结果![]()
步骤2 将![]() 分配权重
分配权重![]() 进行乘加求和,而后对每一个类别的概率图谱根据标签兼容性矩阵μ(m)(xi,l)进行转换更新得到点对能量势函数φp(xi,l);
进行乘加求和,而后对每一个类别的概率图谱根据标签兼容性矩阵μ(m)(xi,l)进行转换更新得到点对能量势函数φp(xi,l);
步骤3 对FCNN- 4s网络的输出求取一元能量势函数φu(xi),然后整合步骤2中的点对能量势函数φp(xi,l)求取整个能量函数![]()
步骤4 将![]() 归一化求取像素归属标签的概率值Qi(xi),并按最大概率取该像素所对应的标签,直到每一个像素点归属于各自标签的概率值迭代达到90%以上退出算法循环,否则将当前计算的像素损失反向传播回FCNN- 4s算法中继续网络参数的学习与修正。
归一化求取像素归属标签的概率值Qi(xi),并按最大概率取该像素所对应的标签,直到每一个像素点归属于各自标签的概率值迭代达到90%以上退出算法循环,否则将当前计算的像素损失反向传播回FCNN- 4s算法中继续网络参数的学习与修正。
算法的伪代码如下所示,最终形成CRF与FCNN- 4s的融合网络结构,可采用反向传播算法端对端地计算每个像素的损失并更新网络参数权重。
Begin
![]() 为归一化因子
为归一化因子
While not converged Qi(xi)≥90% do
end while
End
4 实验结果与分析
4.1 数据和评价标准
实验采用在线MR脑肿瘤数据图库BRATS 2015[注]https:∥www.smir.ch/BRATS/Start2015(Brain tumor image segmentation benchmark, BRATS),图库中每一例患者的FLAIR、T1、T2和T1C等四种模态MR图像都已配准,即各模态图像按人体标准大脑进行线性对准,像素点相互对应,反映大脑中同一位置的肿瘤情况[7]。各模态MR图像的三维大小均为240×240×155,真值标签为多名专家手工标定的肿瘤结果。实验环境如下:CPU Intel Core i7 3.5GHz,GPU NVIDIA GeForce GTX1070,Ubuntu 16.04 LST 64位操作系统,Caffe深度学习框架。
本文采用相似系数(Dice Similarity Coefficient, DSC),灵敏度(Sensitivity)和阳性预测值(Predictive Positivity Value, PPV)等指标评价脑肿瘤的分割结果。其中相似系数表示实验分割结果与标签之间的相似程度,灵敏度代表实验分割后正确肿瘤点占真值肿瘤点的比例,阳性预测值代表实验分割后正确肿瘤点占分割结果为肿瘤点的比例。表达式如下:
(4)
(5)
(6)
式中P表示本文算法分割结果,T表示专家分割脑肿瘤的标签。
4.2 实验参数的设置与模型的训练
实验随机选取100例脑肿瘤患者的MR图像作为训练集。即首先将大小为240×240×155的四种模态的体素图像进行切片处理,随后利用3.1节的图像预处理技术和数据增强技术得到数量为51000、大小为240×240的灰度图像融合训练数据。本文主要对脑肿瘤图像做水平翻转、上下翻转以及逆时针旋转90°、180°和270°的数据增强处理,以提高训练模型的精度。
在训练阶段,使用较为成熟的Caffe深度学习框架对训练集进行模型参数的学习,考虑到Sigmoid、Tanh激活函数容易造成梯度消失,网络选取ReLU作为激活函数,采用常规的SoftMax分类损失函数。由于学习率的选取对网络至关重要,它决定网络是否能够收敛和收敛的程度,既不能太大也不能过小,本文利用原始训练集比较不同学习率下网络的收敛情况,图4反映出当网络选取10e- 08和10e- 09等较大学习率时,会导致训练的损失无法收敛,当学习率为10e-10时,训练损失可以收敛,且比10e-11等小学习率收敛程度更好,所以本文最终采用10e-10的学习率。
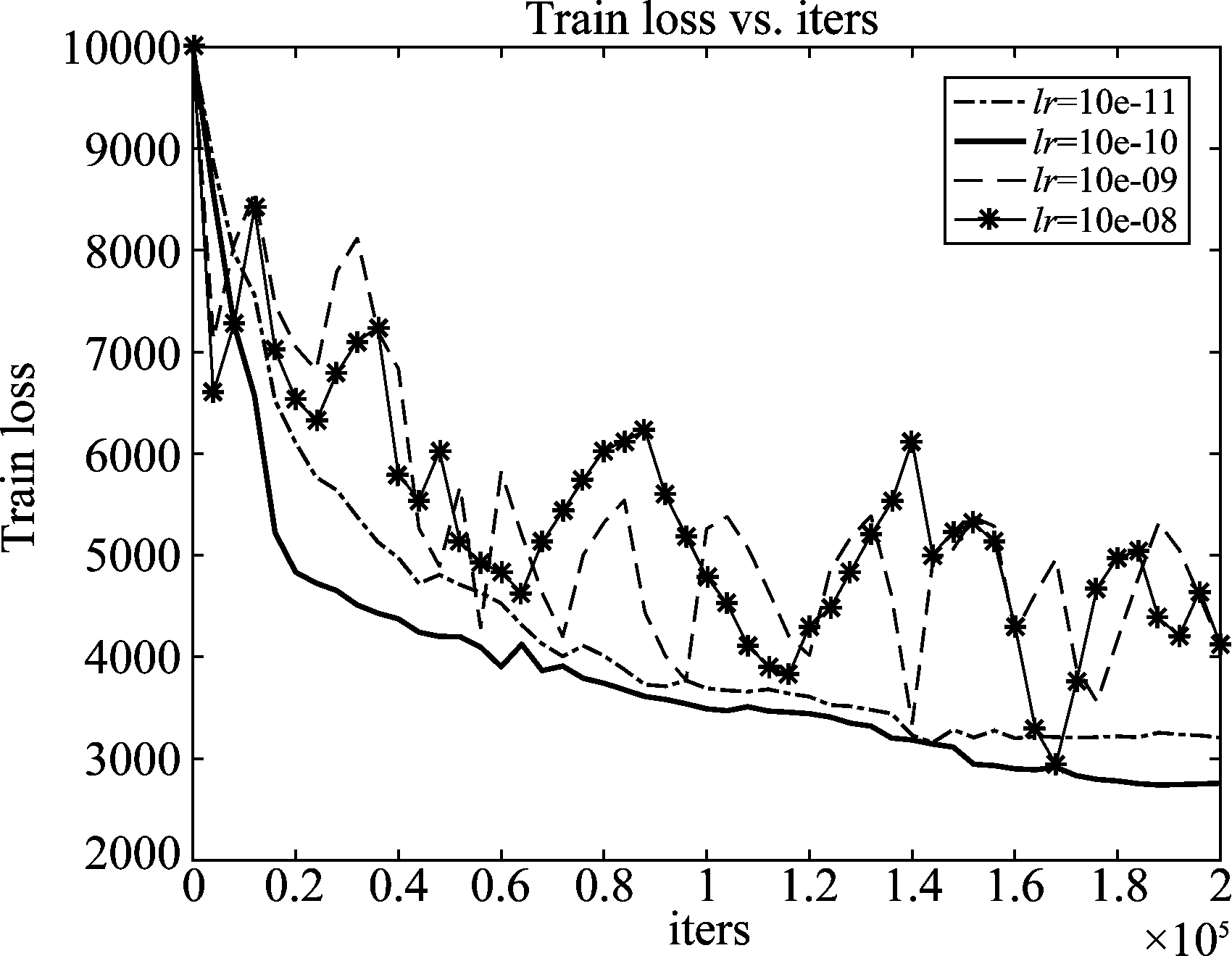
图4 不同学习率下网络的收敛程度
Fig.4 The convergence degree of the network with different learning rates
权重衰减系数对网络的过拟合程度有一定的影响,本文比较不同权重衰减系数下网络的训练准确率,从表2可看出:当权重衰减系数过小时没有起到权重衰减的作用使得网络训练的准确率较差;当权重衰减系数过大时网络出现一定的过拟合,训练准确率有所下降,所以本文最终采取权重衰减系数为0.0005。
表2 不同权重衰减系数下网络的训练准确率
Tab.2 Train accuracy of the network under different weight decay
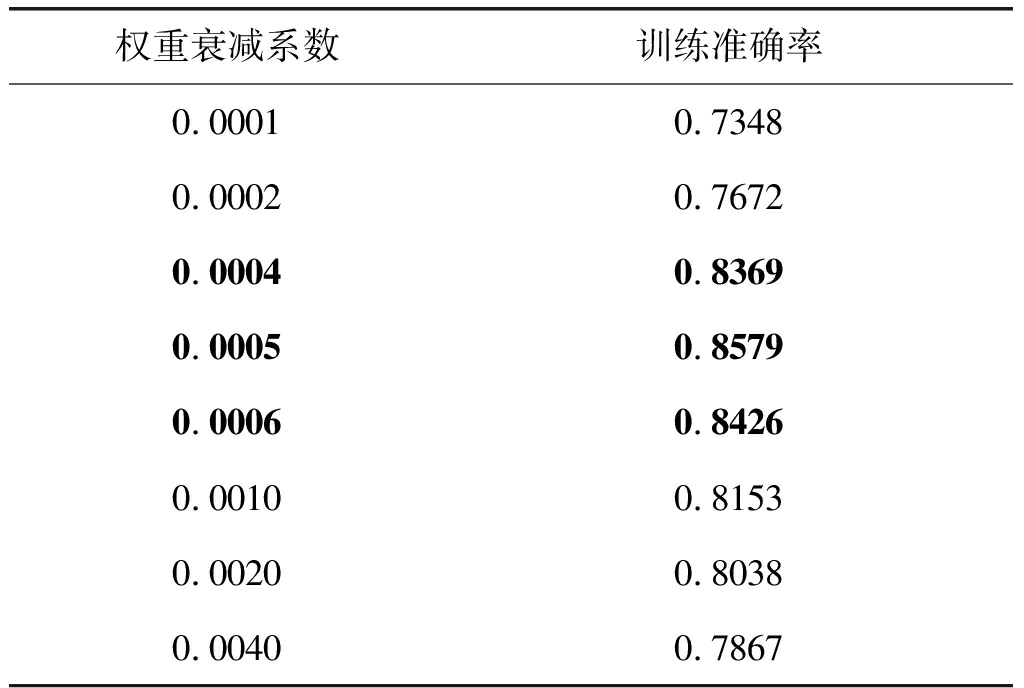
权重衰减系数训练准确率0.00010.73480.00020.76720.00040.83690.00050.85790.00060.84260.00100.81530.00200.80380.00400.7867
为验证加入BN层的有效性,本文利用原始训练集比较加入BN层前后的网络训练损失和训练准确率。图5(a)中加入BN层的网络在迭代8000次后损失已降到5000左右,迭代1.2×105次网络已基本收敛;而无BN层的网络在迭代2.0×104次损失才降到5000左右,迭代近1.6×105次网络才收敛,充分印证了BN层可加速网络的收敛速度,且收敛的损失值较小,图5(b)反映了BN层的加入在一定程度上也提高了训练模型的精度。
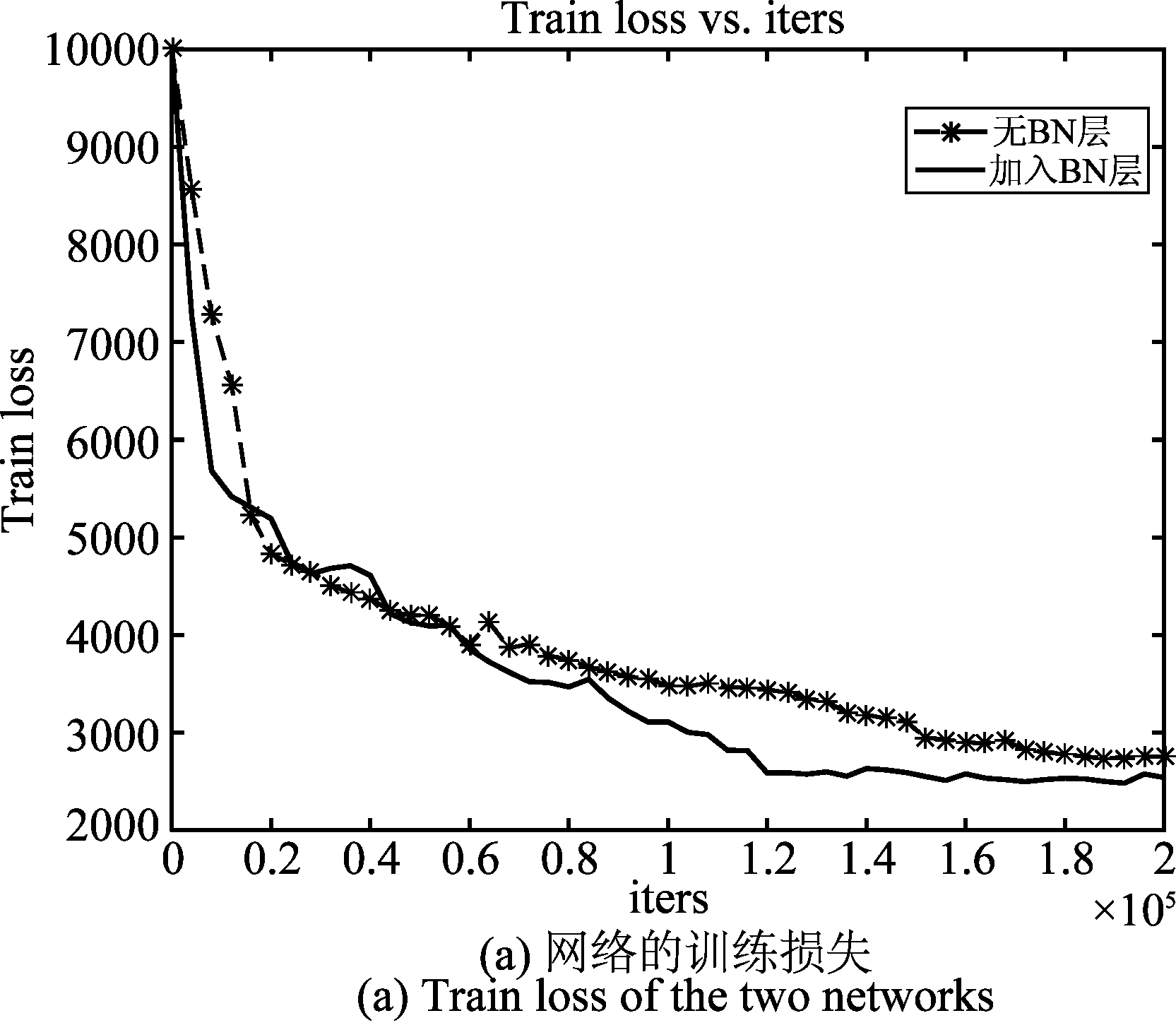
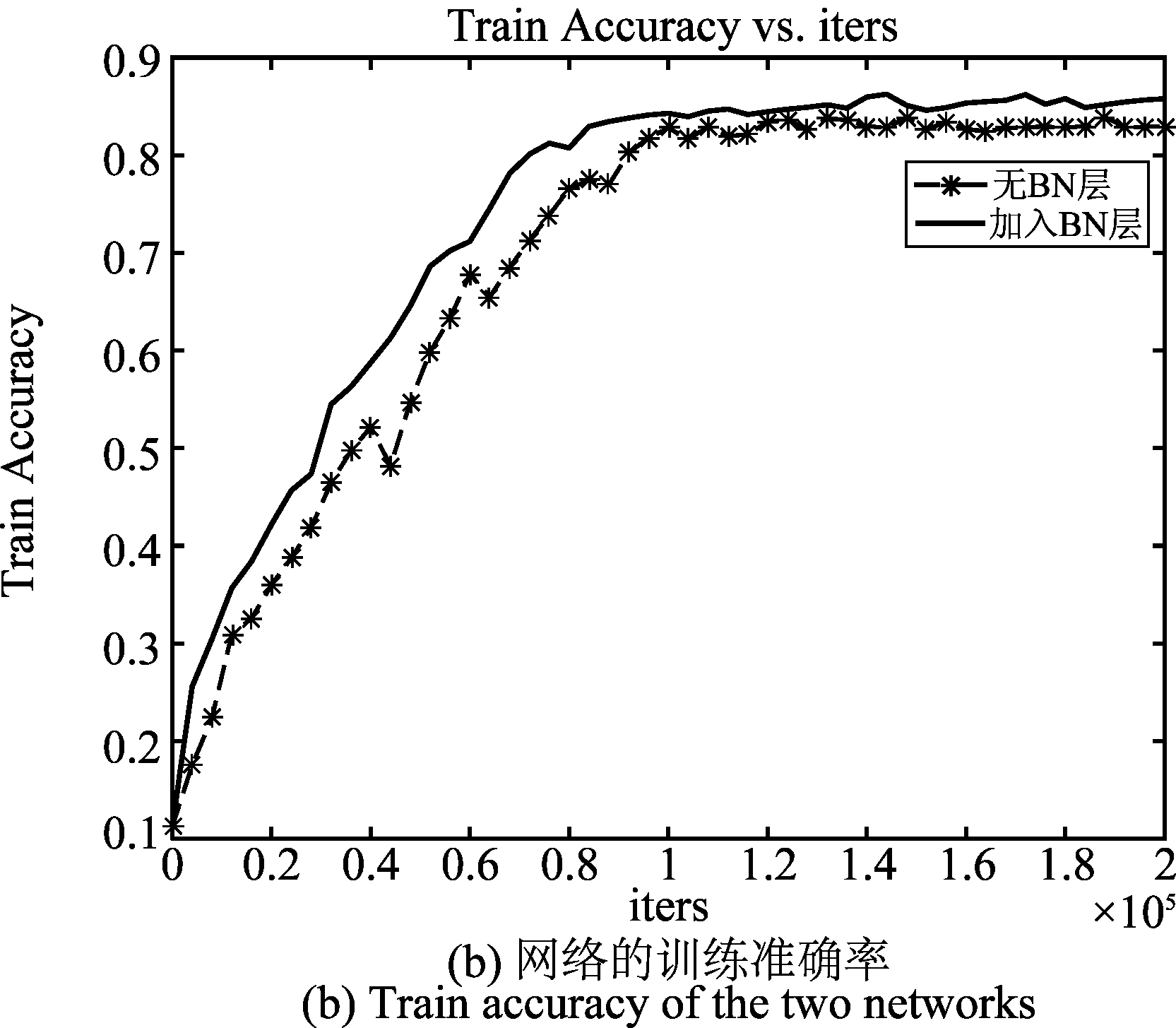
图5 有无BN层的网络训练损失和准确率
Fig.5 Train loss and accuracy of the network whether adding the BN layer
当网络结构和参数正确选择后,为验证扩增的数据集可提高模型精度,本文对不同数据集进行肿瘤分类正确率的比较,从表3可看出,随着数据集的一步步扩增,训练的准确率大幅度提升10.45%,可见增大训练样本可防止网络过拟合,提高模型的泛化能力,这样也保证了训练的模型在测试集上能够实现较好的脑肿瘤分割。
表3 不同数据集下网络的训练准确率
Tab.3 Train accuracy of the network under different data sets
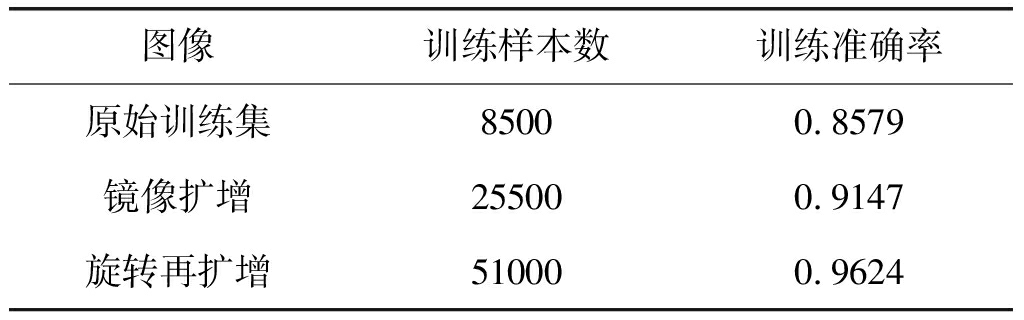
图像训练样本数训练准确率原始训练集85000.8579镜像扩增255000.9147旋转再扩增510000.9624
最后为了验证改进的FCNN- 4s和CRF的融合网络更加有利于肿瘤边界特征提取,本文利用扩增的训练集和以上参数训练了融合两次特征的FCNN- 8s网络和融合三次特征的FCNN- 4s网络。图6表示了各网络所提取的最终脑肿瘤特征信息,黄色程度越深,代表是脑肿瘤的概率越大,蓝色程度越深则为背景的概率越大。从中可看出图6(b)融合更多特征信息的FCNN- 4s网络比图6(a)FCNN- 8s网络分割的脑肿瘤总体轮廓更加明显,而图6(c)通过CRF模型加强了边界信息的提取,使得提取的脑肿瘤特征非常接近标签值。
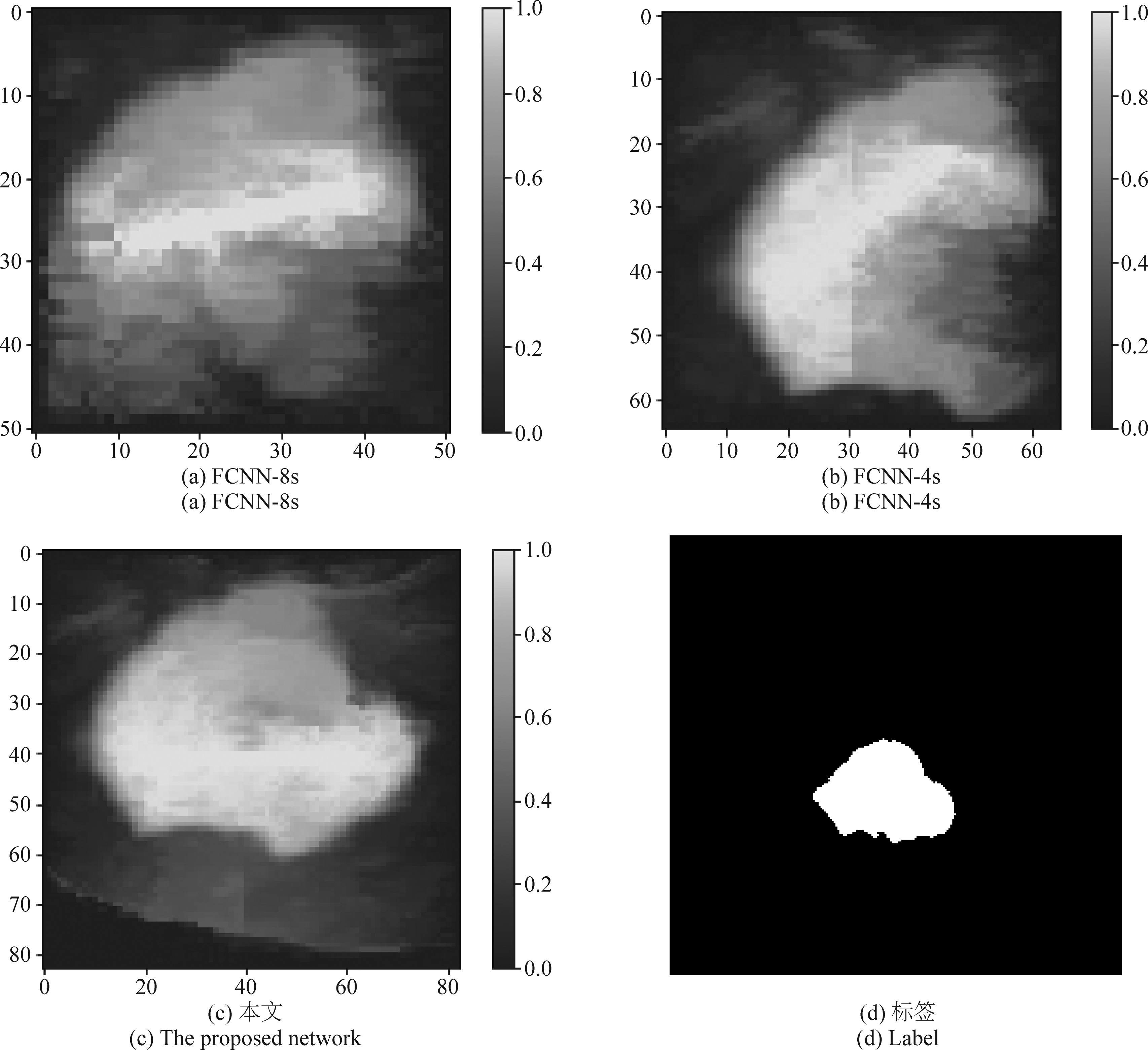
图6 不同网络结构提取的脑肿瘤特征
Fig.6 The brain tumor feature of different networks
4.3 单模态图像与融合模态图像的分割结果比较
实验同时选取50例脑肿瘤患者的MR图像进行模型测试。为了比较本文提出的算法在单模态图像与融合图像分割效果上的差异,选取脑肿瘤边界较明显的FLAIR单模态图像,与采用灰度图像融合的预处理图像进行分割结果上的比较。从实验结果图7中可看出,当用本文算法分割FLAIR单模态图像时,可正确分割出脑肿瘤的总体轮廓,但非肿瘤区域存在大量的散点,即出现过分割现象;当分割灰度融合模态图像时,由于三种模态的肿瘤信息可以相互补充,加强了边界等特征的提取,使得脑肿瘤的轮廓不仅被正确分割出来,在一定程度上也减少了过分割的现象,分割的肿瘤边界也更加精细。
随后采用三种指标定量分析分割结果的好坏,从表4可看出,多模态融合图像相对单模态图像在相似性指数、灵敏度以及阳性预测率等分割精度上都有了较大提升,尤其是Dice提高了6.33%。
表4 算法在单模态与融合模态图像上的分割性能评估
Tab.4 Segmentation performance evaluation of algorithm in single mode and fused mode
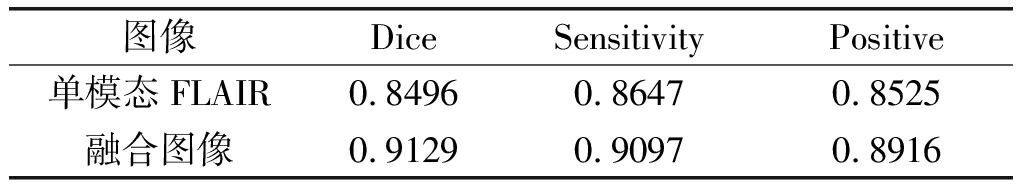
图像DiceSensitivityPositive单模态FLAIR0.84960.86470.8525融合图像0.91290.90970.8916
4.4 对比和讨论
为了验证所提改进算法的优越性,本文与只进行两次特征融合的简单分割算法FCNN- 8s以及没有融合CRF的FCNN- 4s算法进行比较,同时实验还与著名学者Havaei[10]和Pereira[11]等提出的传统CNN算法进行对比,从图8可看出,Havaei[10]算法存在明显的过分割,分割的脑肿瘤边界不明显且存在较多的孤立散点;Pereira[11]算法由于网络层数更深,使得分割模型的精度更好,性能整体较Havaei[10]算法略有提升,并在一定程度上减少了脑肿瘤的过分割;FCNN- 8s虽然获得了平滑的脑肿瘤分割轮廓,但由于特征融合较少导致脑肿瘤边界不够精细且分割精度不高;FCNN- 4s相对FCNN- 8s来说在获取脑肿瘤边界信息上有一定增强,但分割出的脑肿瘤边界还是不够细腻。尤其在分割第3例复杂脑肿瘤图像时,由于肿瘤边界太复杂导致各种算法的分割结果都差强人意,但采用本文算法却能够分割出比较满意的结果。总的来说,采用特征融合更加精细的FCNN- 4s模型,且融合CRF形成的端对端的算法结构能够使得相似的像素点获得相同的标签,从而细化脑肿瘤的边界,有效解决脑肿瘤的过分割与欠分割问题。
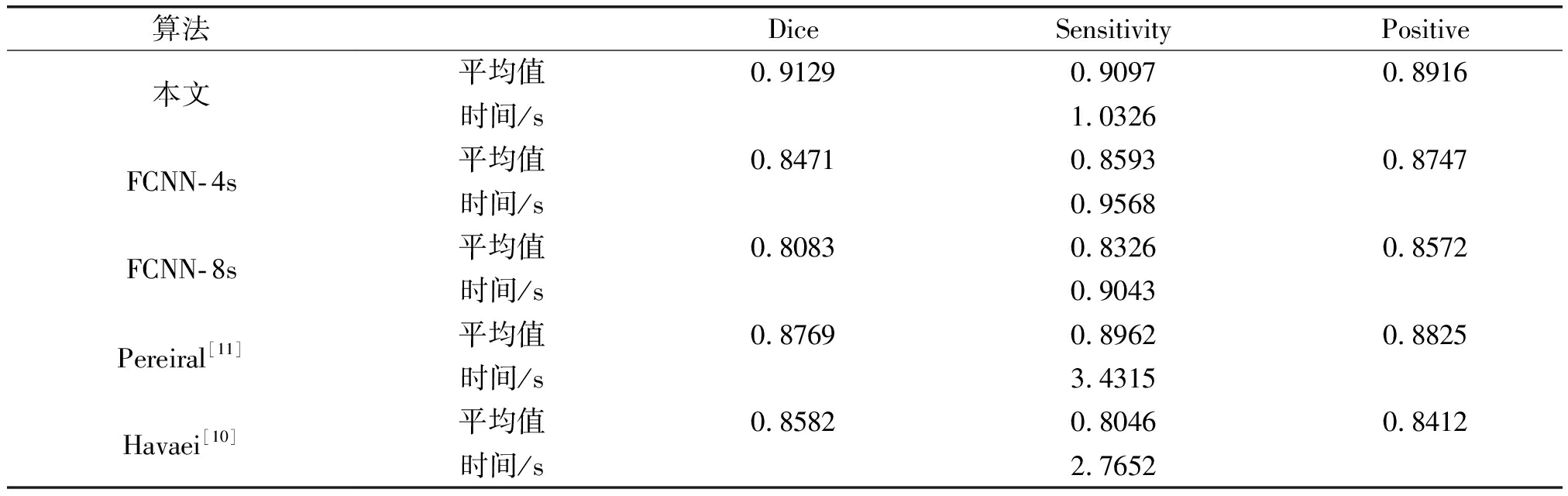
另外从表5中也可看出本文算法相对其他算法具有较高的分割精度,Dice较两位学者提出的算法分别提高了5.47%和3.60%,较无融合CRF的FCNN算法更是平均提高了8.52%。再从分割一幅脑肿瘤图像的平均时间来看,本算法在预测阶段具有较高的实时性,平均1 s内便可完成一幅脑肿瘤图像的分割。
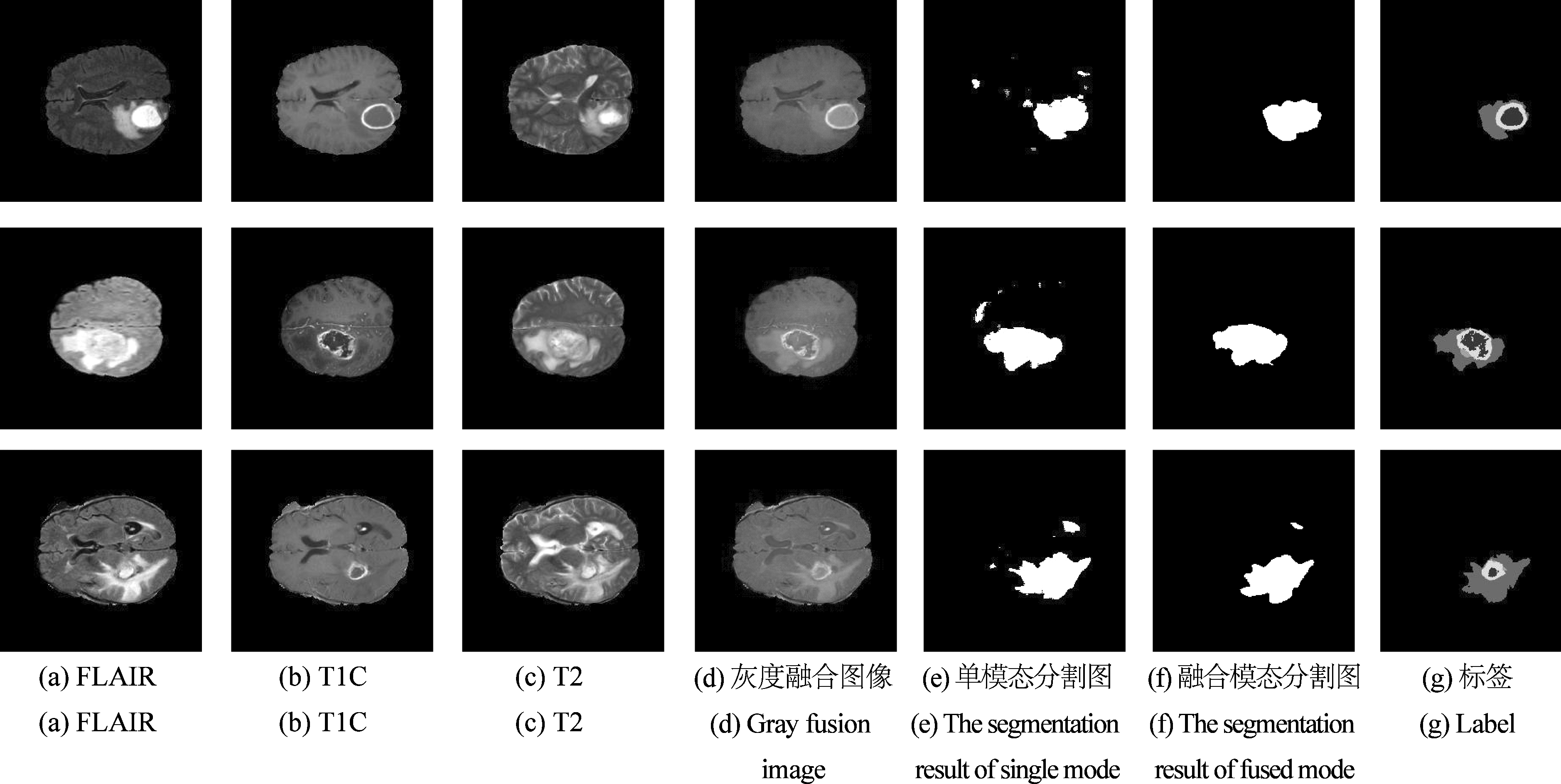
图7 算法在单模态与融合模态图像上的分割结果比较图
Fig.7 Compared with the segmentation results between single mode and fused mode image
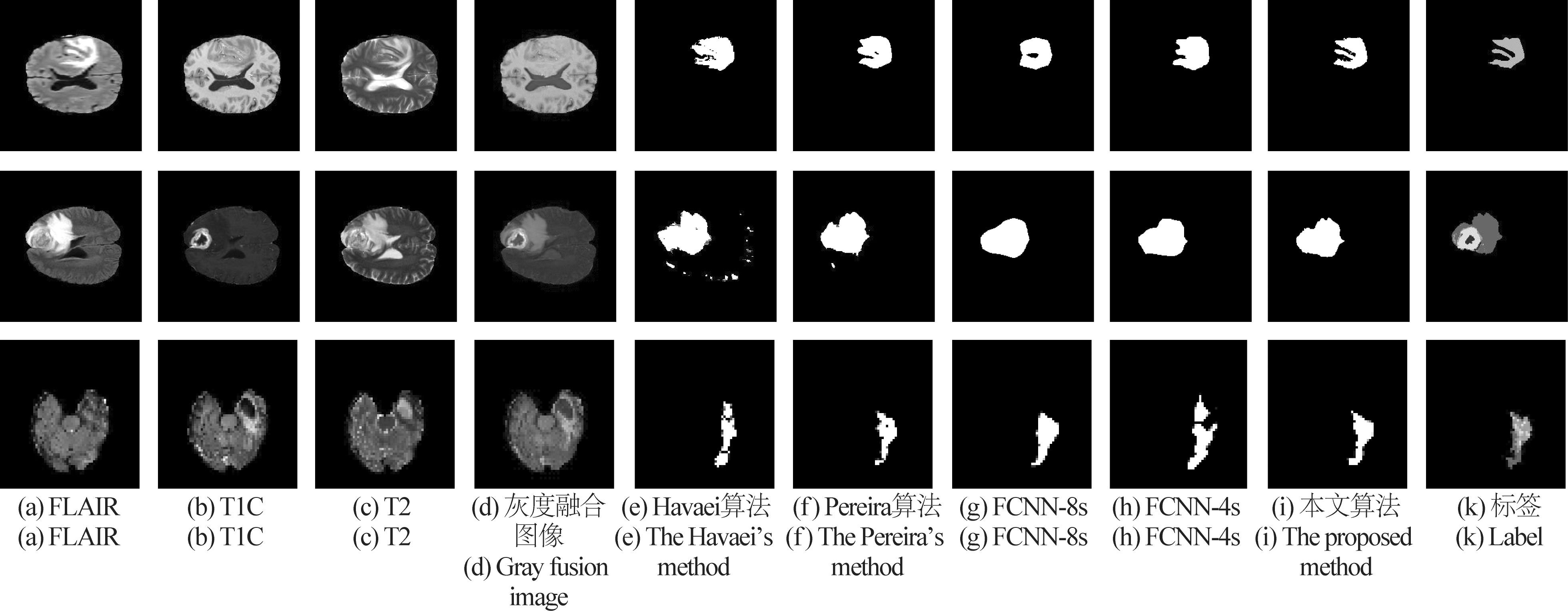
图8 五种算法的分割结果比较图
Fig.8 Compared with the segmentation results of five segmentation methods
表5 五种算法的分割性能评估
Tab.5 Segmentation performance evaluation of five methods
算法DiceSensitivityPositive本文平均值0.91290.90970.8916时间/s1.0326FCNN-4s平均值0.84710.85930.8747时间/s0.9568FCNN-8s平均值0.80830.83260.8572时间/s0.9043Pereiral[11]平均值0.87690.89620.8825时间/s3.4315Havaei[10]平均值0.85820.80460.8412时间/s2.7652
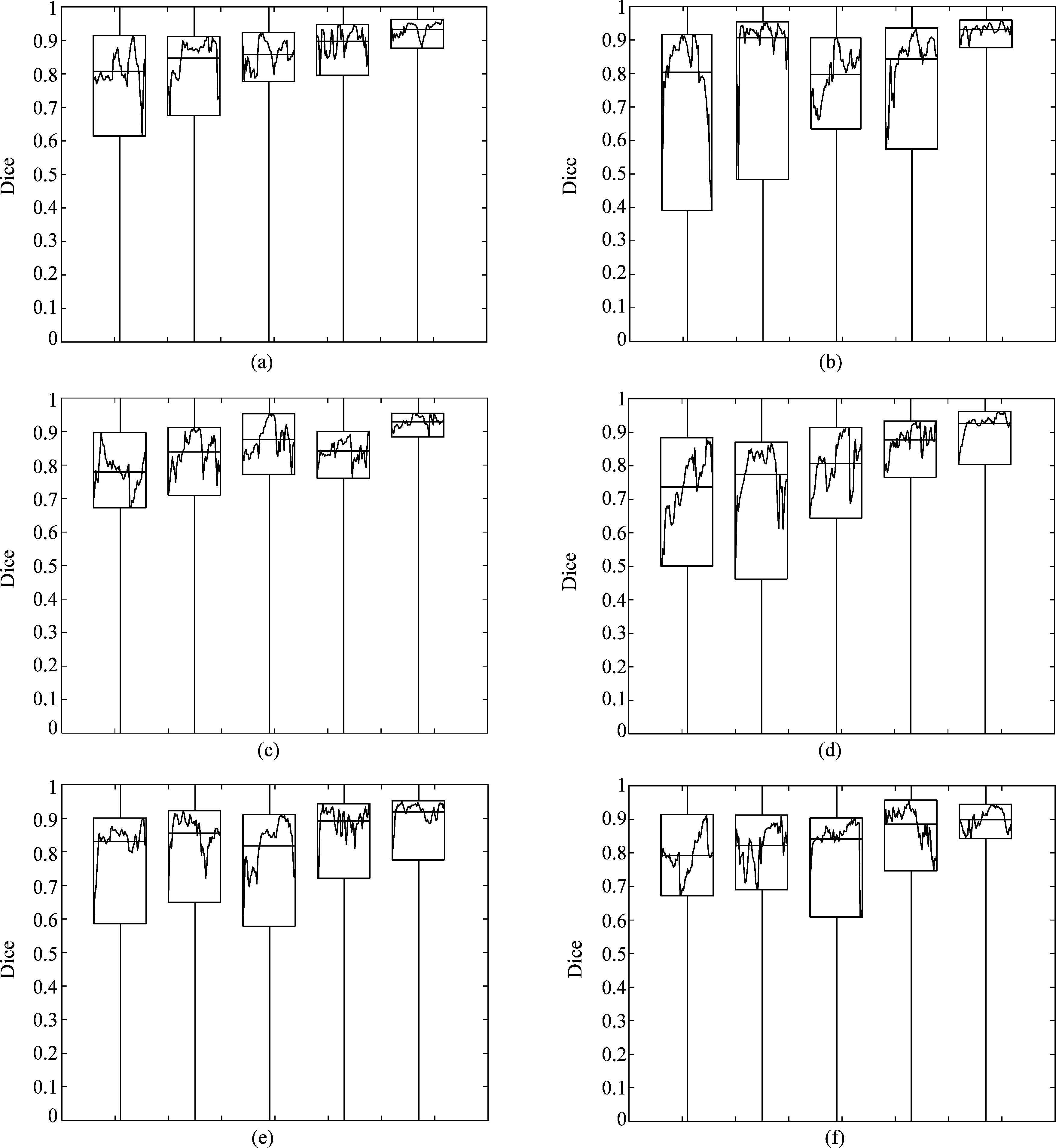
图9 五种算法的稳定性评价图
Fig.9 The stability evaluation of five algorithms
同时为了验证本文算法的稳定性,实验随机选取6例患者每例各50幅脑肿瘤切片图像,对每个算法进行Dice评价。图9中每幅图像代表一个患者采用不同算法分割的Dice值,每个小矩形框表示一个算法,从左到右依次为FCNN- 8s算法,FCNN- 4s算法,Havaei[10]算法,Pereiral[11]算法以及本文算法。矩形框的高度反映了算法的稳定性,矩形框越高则说明算法Dice波动太大,算法稳定性较差;矩形框内的横线为该算法平均Dice值。可看出五种算法的分割精度基本都达到80%以上,但FCNN- 8s算法不论是在分割精度还是在稳定性上表现都较差;Pereiral[11]算法较Havaei[10]算法在分割精度上略有提升,稳定性也较好;FCNN- 4s算法较FCNN- 8s算法分割性能大幅度提高,稳定性相对两位学者较差。但融合CRF和BN的本文算法在分割脑肿瘤时不仅精度大幅度提高,平均Dice高达91.29%左右,而且还具有较高的稳定性,整体性能优于其他算法。
5 结论
本文针对卷积神经网络分割脑肿瘤计算复杂度高且精度不高的缺陷提出了一种改进的脑肿瘤图像分割算法。首先利用改进的全卷积神经网络实现脑肿瘤像素级别的粗分割,而后基于图像标签之间的相关性,融合概率图谱模型条件随机场,形成可训练的端对端的分割算法。与传统的卷积神经网络分割算法相比,本文的算法结构更加精细,能有效解决脑肿瘤的过分割与欠分割问题,分割预测实时性更好,稳定性较强,可实现较高的分割精度。
[1] Schwartzbaum J A, Fisher J L, Aldape K D, et al. Epidemiology and molecular pathology of glioma[J]. Nature Clinical Practice Neurology, 2006, 2(9):494-503.
[2] Menze B, Reyes M, Van L K. The Multimodal Brain Tumor Image Segmentation Benchmark (BRATS)[J]. IEEE Transactions on Medical Imaging, 2015, 34(10):1993-2024.
[3] Mazzara G P, Velthuizen R P, Pearlman J L, et al. Brain tumor target volume determination for radiation treatment planning through automated MRI segmentation[J]. International Journal of Radiation Oncology Biology Physics, 2004, 59(1):300-312.
[4] Bauer S, Wiest R, Nolte L P, et al. A survey of MRI-based medical image analysis for brain tumor studies[J]. Physics in Medicine & Biology, 2013, 58(13):97-129.
[5] Levine M D,Shaheen S I. A modular computer vision system for picture segmentation and interpretation[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 1981, 3(5):540-556.
[6] Szil gyi L, Lefkovits L, Benyó B. Automatic Brain Tumor Segmentation in multispectral MRI volumes using a fuzzy c-means cascade algorithm[C]∥International Conference on Fuzzy Systems and Knowledge Discovery. IEEE,2016:174-181.
gyi L, Lefkovits L, Benyó B. Automatic Brain Tumor Segmentation in multispectral MRI volumes using a fuzzy c-means cascade algorithm[C]∥International Conference on Fuzzy Systems and Knowledge Discovery. IEEE,2016:174-181.
[7] 童云飞, 李锵, 关欣. 改进的多模式脑肿瘤图像混合分割算法[J]. 信号处理, 2018,34(3):340-346.
Tong Yunfei, Li Qiang, Guan Xin. An improved multi-modal brain tumor segmentation hybrid algorithm[J]. Journal of Signal Processing, 2018,34(3):340-346.(in Chinese)
[8] Soltaninejad M, Yang G, Lambrou T, et al. Automated brain tumour detection and segmentation using superpixel-based extremely randomized trees in FLAIR MRI[J]. International Journal of Computer Assisted Radiology & Surgery, 2016, 12(2):183-203.
[9] Fathima M M, Manimegalai D, Thaiyalnayaki S. Automatic detection of tumor subtype in mammograms based on GLCM and DWT features using SVM[C]∥International Conference on Information Communication and Embedded Systems. IEEE, 2013:809- 813.
[10] Havaei M, Davy A, Warde-Farley D, et al. Brain tumor segmentation with Deep Neural Networks[J]. Medical Image Analysis, 2015, 35:18-31.
[11] Pereira S, Pinto A, Alves V, et al. Brain Tumor Segmentation Using Convolutional Neural Networks in MRI Images[J]. IEEE Transactions on Medical Imaging, 2016, 35(5):1240-1251.
[12] 师冬丽, 李锵, 关欣. 结合卷积神经网络和模糊系统的脑肿瘤分割[J]. 计算机科学与探索, 2018,12(4):608- 617.
Shi Dongli, Li Qiang, Guan Xin. Brain tumor image segmentation algorithm based on convolution neural network and fuzzy inference system[J]. Journal of Frontiers of Computer Science and Technology, 2018,12(4):608- 617.(in Chinese)
[13] Liu Z, Li X, Lu P, et al. Semantic image segmentation via deep parsing network[C]∥IEEE International Conference on Computer Vision. IEEE, 2015:1377-1385.
[14] Chen L C, Papandreou G, Kokkinos I, et al. Semantic Image Segmentation with Deep Convolutional Nets and Fully Connected CRFs[J]. Computer Science, 2014(4):357-361.
[15] Zheng S,Jayasumana S, Romera-Paredes B, et al. Conditional Random Fields as Recurrent Neural Networks[C]∥IEEE International Conference on Computer Vision. IEEE, 2016:1529-1537.
[16] Lécun Y, Bottou L, Bengio Y, et al. Gradient-based learning applied to document recognition[J]. Proceedings of the IEEE, 1998, 86(11):2278-2324.
[17] Krizhevsky A, Sutskever I, Hinton G E. ImageNet classification with deep convolutional neural networks[C]∥International Conference on Neural Information Processing Systems. Curran Associates Inc. 2012:1097-1105.
[18] Long J,Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation[C]∥Computer Vision and Pattern Recognition. IEEE, 2015:3431-3440.
[19] Ioffe S, Szegedy C. Batch normalization: accelerating deep network training by reducing internal covariate shift[C]∥International Conference on International Conference on Machine Learning. JMLR.org, 2015:448- 456.


